Predicting the spatial and temporal variations in habitat characteristics of Harpadon nehereus in the Changjiang River Estuary based on ensemble modeling
-
摘要: 为了评估“集成方法”能否改进物种分布模型(Species Distribution Models, SDMs)在海洋环境高度动态变化的河口区域的预测性能,本研究基于2013–2021年长江口海洋生物资源调查数据,使用8种基于不同算法的单一模型对长江口的优势物种之一龙头鱼(Harpadon nehereus)构建了栖息地生境的集成模型(Ensemble Model, EM)。结果显示:(1)所有单一模型的预测性能均优于随机分布模型,而EM具有最高的预测准确性和稳健性(受试者工作特征曲线下面积(Area Under receiver operating character Curve, AUC)= 0.875;真实技巧统计值(True skill statistic, TSS)= 0.650;KAPPA系数= 0.560;总体精度(Overall accuracy, OA)= 0.867);(2)EM能最为准确地识别出龙头鱼的出现点和未出现点,也能清晰地区分出未采样区域适宜性水平的差异,并预测出不同模型共同的高适宜区域;(3)最后,EM能准确识别龙头鱼的关键环境需求并反映出多模型集中的变化趋势,其最适盐度、温度和化学需氧量的范围分别为2.754~30.300、28.278~30.934℃、4.605~8.080 mg/L。本研究可为长江口龙头鱼资源的可持续利用和栖息地保护工作提供更可靠的研究方法。Abstract: To evaluate whether the “ensemble approach” can enhance the predictive performance of Species Distribution Models (SDMs) in dynamically changing estuarine environments, this study utilized eight single models based on different algorithms to construct an Ensemble Model (EM) for the habitat of Harpadon nehereus, a dominant species in the Changjiang River Estuary. The data used for modeling were derived from marine biological resource surveys conducted in the Changjiang River Estuary from 2013 to 2021. The results showed that: (1) All single models outperformed the random distribution model, with the EM demonstrating the highest predictive accuracy and robustness (Area Under receiver operating character Curve, AUC = 0.875; True skill statistic, TSS = 0.650; KAPPA = 0.560; Overall accuracy, OA = 0.867). (2) The EM accurately identified both presence and absence stations of H. nehereus, clearly differentiated suitability levels in unsampled regions, and predicted areas of high suitability shared by different models. (3) Finally, the EM accurately identified the key environmental requirements of H. nehereus and reflected the central tendency across multiple models. The most suitable habitat for H. nehereus was found in waters with salinity, temperature, and chemical oxygen demand ranges of 2.754−30.300, 28.278−30.934℃, and 4.605−8.080 mg/L, respectively. This study provides a more reliable research method for the sustainable utilization and habitat protection of H. nehereus resources in the Changjiang River Estuary.
-
1. 引言
长江口是西太平洋最大的河口,区域内滩槽交错,水系动态互动频繁,支撑了丰富的渔业资源和复杂的水生生物群落结构[1]。然而,由于过度捕捞、环境污染、工程开发等人类活动的胁迫,长江口生态系统的结构特征已逐步发生变化[2],表现在多种传统优质的鱼类种群规模大幅减小,小型鱼类的资源量不断增大[3]。龙头鱼(Harpadon nehereus)作为一种典型代表性的小型鱼类,已成为了长江口的优势物种之一[4]。因此,加强对龙头鱼物种分布和资源动态的监测和研究,不仅有助于理解长江口水生生物群落结构的演替过程,也有助于制定合理的养护管理措施来维持生态系统的稳定性。
物种分布模型(Species Distribution Models, SDMs)已经成为渔业生态学家预测和管理海洋生物资源栖息地分布的重要工具[5],基于不同算法规则、不同类型的模型被广泛用于物种潜在生境的预测。绝大多数鱼类栖息地建模过程依赖于“单一模型”,即仅基于一种算法构建的SDMs[6–7]。然而,单一模型对建模方法的假设和具体特征十分敏感[8]。至今,针对长江口龙头鱼开展的研究十分有限,仅有少部分研究基于单一模型探究了龙头鱼的时空分布特征和栖息地偏好[4, 9]。
SDMs的“集成”方法正成为生态学家解决生态学问题的一种流行方法,当多种模型具有近似等效的预测性能时,集成预测使得建模者无需在这些模型中选择一个效果最佳模型,并且还可减少因过度拟合而可能产生的误差,从而使得集成建模方法在预测物种—环境关系方面优于单一建模方法[10]。但是,并非所有研究都支持集成模型(Ensemble Model, EM)的预测表现总是优于单一模型[11]。因此,针对具体的目标物种和研究区域,需要专门的研究来评估EM与各单一模型性能相比表现是否更加优异,从而获得理解物种的环境需求和模拟栖息地适宜性的最佳工具。
本研究将以长江口的龙头鱼作为研究对象,使用包括机器学习算法、分类算法、回归算法和包络算法在内的4类常用算法,构建8种单一模型,并将它们集成开发出1种EM,通过评估EM能否提供比单一模型更佳的预测性能,为长江口龙头鱼资源的可持续利用和水生生态系统的保护管理确定更加可靠的科学工具。
2. 材料和方法
2.1 研究区域概况
长江口位于长江径流与近岸潮流、淡水与咸水相交汇的区域,是典型的河口生态区[12],为许多水生生物提供了重要的“三场一通道”的生态功能[13]。由于受到长江径流、台湾暖流、黄海沿岸流等水体的影响[14],长江口的盐度、水温和营养盐等理化特征因长江径流量的变化而产生显著的季节性变化[14–16]。在夏、秋季,长江冲淡水阻止了高盐水的上溯和向南支的倒灌,促使南支水域呈现出明显的低盐特征[17];同时,以高温形式向外海扩展的长江径流使得水域内平均最高水温达到27.5~38.8℃[14–15];在冬、春季,高盐水随涨潮流上溯至北支上游,使水体盐度基本保持在20以上[17],平均水温跌至全年最低的5.6~6.7℃[14–15]。本研究覆盖的地理范围为31°14′48″~31°45′00″N,121°19′12″~122°17′31.2″E;(图1),是长江径流由淡水转入咸水的典型过渡区域。
2.2 数据来源
本研究数据来源于2013–2021年上海市水生野生动植物保护研究中心开展的长江口海洋生物资源监测工作,调查时间为每年的5月(春季)、8月(夏季)、11月(秋季)和2月(冬季),站点图如图1所示。使用底拖网进行拖曳调查,使用的网具网口高2 m,网口宽6 m,网长10 m,纲长6 m,囊网网目20 mm,在每个站点以2 kn的航速拖曳0.5 h(图1)。拖曳结束后将渔获物带至实验室进行种类鉴定、计数和称重,以获得各站点的种类组成。若在某站点监测到龙头鱼,则记为出现点;反之则记为未出现点,将具有空间信息的龙头鱼出现及未出现数据作为物种数据用于模型构建。
根据先前对河口鱼类关键生境需求分析的资料[18–20],本研究选择水温(temperature, Tem)、盐度(salinity, Sal)、溶解氧(dissolved oxygen, DO)、化学需氧量(chemical oxygen demand, COD)、pH值、叶绿素a(chlorophyll a, Chl a)和水深(Depth)作为龙头鱼栖息地所需的潜在环境需求。
根据《海洋调查规范(GB/T 12763.6–2007)》[21]和《渔业水质标准(GB 11607–89)》[22]的要求,使用多参数水质分析仪(WTW Multi-3430)对调查站点海表的Sal、Tem、DO、pH值进行现场测定,通过电子海图显示的位置水深进行Depth记录。使用有机玻璃采水器在各站点采取500 mL水样,通过0.45 µm混合纤维素酯滤膜对水样进行减压抽滤,将截留浮游植物细胞的滤膜置于离心管中,通过低温保存运回实验室后使用紫外分光光度计测定Chl a含量;并使用重铬酸盐法对水样的COD含量进行测定[23]。
此外,河口区域作为许多鱼类良好的索饵场[24],饵料生物的资源密度也被视为影响鱼类空间分布的重要因素[24–25]。根据长江口水生动物食物网营养结构的分析结果,葛氏长臂虾(Palaemon gravieri)和巨指长臂虾(Palaemon macrodactylus)是龙头鱼捕食的两种重要饵料生物[26]。因此,本研究选择葛氏长臂虾和巨指长臂虾的资源密度总和作为影响龙头鱼出现概率的生物性变量[24],其计算方法如下式所示[27]:
$$ {{\mathrm{prey}}_{{s}}} = \frac{{{U_s}}}{{{T_s}}}, $$ (1) 式中:preys为第s个调查站点的饵料生物资源密度(ind/h),Us为在第s个调查站点作业获得的葛氏长臂虾和巨指长臂虾的渔获物尾数(ind);Ts为在第s个调查站点的拖曳时间(h)。
2.3 环境变量的选择和插值
采用皮尔逊相关性系数分析对所有环境数据进行两两配对分析,避免由于变量间的相关性而造成的统计信息冗余和预测结果的过拟合[28]。使用含障碍的样条函数工具(Spline with Barriers)对调查站点的各项环境数据进行插值,并将研究区域裁剪至相同范围,栅格大小重采样至1′×1′。最终的EM及各单一模型的栖息地分布预测结果被转换为ASCII格式,输入至ArcGIS中实现可视化。上述环境变量相关性分析和插值过程分别在Origin2022和ArcGIS10.4.1中实现。
2.4 EM模型建模、验证及评估
2.4.1 模型的交叉验证及评估
本研究进行100次5折交叉验证,4/5的数据作为训练集以训练模型,1/5的数据作为测试集以进行单一模型的预测和评估;采用真实技巧统计值(True skill statistic, TSS)、受试者工作特征曲线下面积(Area Under receiver operating character Curve, AUC)、Kappa系数和总体精度(Overall accuracy, OA)4种指标评价单一模型的预测结果,本研究的建模流程基于R 4.2.0软件中的BIOMOD2 4.2.3软件包实现,各单一模型的具体参数设置见表1。
表 1 本研究中各单一模型的参数设置Table 1. Parameter settings for each single model in this study类别 模型 参数 值 描述 机器学习算法 梯度提升机
(Gradient Boosting Machine, GBM)n.trees 2500 生成树的数量 interaction.depth 7 交互项的最大深度 shrinkage 0.001 在构建每棵树时,收缩节点的权重 随机森林
(Random forest, RF)ntree 500 生成树的数量 mtry default 每次分割时随机抽样作为候选变量的数量 nodesize 5 每个终端节点的最少样本数 人工神经网络
(Artificial Neural Network, ANN)size NULL 神经网络的规模 decay NULL 学习率衰减因子 maxit 200 训练神经网络时的最大迭代次数 分类算法 分类树分析
(Classification Tree Analysis, CTA)minbucket 5 节点中最少的样本数 minsplit 5 最小分裂点的值 cp 0.001 控制修剪范围 柔性判别分析(Flexible Discriminant Analysis, FDA) Method Mars FDA的建模方法 add_args NULL 附加参数列表 回归算法 广义加性模型(Generalized Additive Model, GAM) algo GAM_mgcv 使用的算法和优化方法 k −1 平滑器的阶次 interaction.level 0 交互项的阶次 广义线性模型
(Generalized Linear Model, GLM)type Quadratic 模型类型 interaction.level 0 交互项的阶次 mustart 0.5 默认起始值 包络算法 表面分布区分室模型(Surface Range Envelop, SRE) quant 0.025 模型的分辨率或离散程度 TSS取值范围为[−1, 1],其中1表示模型预测值和观测值完全一致,0或更小的值表示模型表现不如随机[29];AUC取值范围为[0, 1],通常认为AUC值大于0.8的模型其拟合效果良好[30];Kappa系数的值域为[0, 1],该值越高表明模型的性能越好[31];OA衡量了真阳性和真阴性结果在所有预测结果中的出现概率,倾向于对模型性能进行单一且整体的衡量,该值越高说明模型预测结果可信度越高[31–32]。
2.4.2 模型的筛选与集成
多数基于EM的研究使用AUC作为单一模型集成的筛选标准[33],因此本研究在每一组(共5×100 =500组)运行中筛选出AUC值大于0.8的单一模型进行集成,各单一模型和EM的模拟流程详见图2。
 图 2 构建长江口龙头鱼栖息地模型的技术路线图(GBM:梯度提升机;RF:随机森林;ANN:人工神经网络;CTA:分类树分析;FDA:柔性判别分析;GAM:广义加性模型;GLM:广义线性模型;SRE:表面分布区分室模型;EM:集成模型)Figure 2. Technical framework for constructing the habitat model of Harpadon nehereus in the Changjiang River Estuary (GBM: Gradient Boosting Machine; RF: Random Forest; ANN: Artificial Neural Network; CTA: Classification Tree Analysis; FDA: Flexible Discriminant Analysis; GAM: Generalized Additive Model; GLM: Generalized Linear Model; SRE: Surface Range Envelop; EM: Ensemble Model)
图 2 构建长江口龙头鱼栖息地模型的技术路线图(GBM:梯度提升机;RF:随机森林;ANN:人工神经网络;CTA:分类树分析;FDA:柔性判别分析;GAM:广义加性模型;GLM:广义线性模型;SRE:表面分布区分室模型;EM:集成模型)Figure 2. Technical framework for constructing the habitat model of Harpadon nehereus in the Changjiang River Estuary (GBM: Gradient Boosting Machine; RF: Random Forest; ANN: Artificial Neural Network; CTA: Classification Tree Analysis; FDA: Flexible Discriminant Analysis; GAM: Generalized Additive Model; GLM: Generalized Linear Model; SRE: Surface Range Envelop; EM: Ensemble Model)选择加权平均投票法对单一模型的输出结果进行整合,具体方法为根据每组单一模型的AUC得分对模型进行加权,各单一模型所占权重的计算方法如下式所示[34]:
$$ {\mathrm{W{A}}_{ji}} = \frac{{{\mathrm{AU{C}}_{ji}} - {\mathrm{Min}}({\mathrm{AU{C}}_i})}}{{{\mathrm{Max}}({\mathrm{AU{C}}_i}) - {\mathrm{Min}}({\mathrm{AU{C}}_i})}}, $$ (2) 式中:WAji为EM第i次预测中,被纳入EM建模的单一模型j所占的权重;AUCji为EM第i次预测中,单一模型j的AUC得分。
单一模型的评价得分越高,其所占权重就越大[35]。本研究的EM按照单一模型所占权重来预测物种出现概率,计算方法如下式所示[36]:
$$ {\mathrm{E{M}}_{si}} = \dfrac{1}{{\sum\limits_{j = 1}^n {{\mathrm{W{A}}_{ji}}} }}\sum\limits_{j = 1}^n {{\mathrm{W{A}}_{ji}} \cdot mjsi}, $$ (3) 式中:EMsi为EM第i次预测栅格单元s中物种的潜在出现概率;WAji为EM第i次预测中,单一模型j所占的权重;mjsi为EM第i次预测中,单一模型j预测的给定栅格单元s中的物种潜在出现概率;n为被纳入EM建模的单一模型的数量。
3. 结果
3.1 变量选择
本研究所选的部分解释变量存在明显的季节间变化,通过皮尔逊相关性系数矩阵分析,候选的解释变量中DO和Tem呈显著负相关(|R| > 0.7)(图3),因此剔除DO,保留剩余的7个变量用于长江口龙头鱼的物种分布模型构建。
 图 3 各解释变量的皮尔逊相关性系数矩阵分析图(COD:化学需氧量;Depth:水深;Tem:水温;Chl a:叶绿素a;Sal:盐度;DO:溶解氧;prey:饵料生物资源密度)Figure 3. Pearson correlation coefficient for explanatory variables (COD: chemical oxygen demand; Tem: temperature; Chl a: chlorophyll a; Sal: salinity; DO: dissolved oxygen; prey: the stock density of prey species)
图 3 各解释变量的皮尔逊相关性系数矩阵分析图(COD:化学需氧量;Depth:水深;Tem:水温;Chl a:叶绿素a;Sal:盐度;DO:溶解氧;prey:饵料生物资源密度)Figure 3. Pearson correlation coefficient for explanatory variables (COD: chemical oxygen demand; Tem: temperature; Chl a: chlorophyll a; Sal: salinity; DO: dissolved oxygen; prey: the stock density of prey species)3.2 单一模型与EM的性能比较
在所选的8种单一模型中,7种单一模型(RF、GBM、GAM、CTA、FDA、ANN和GLM)中符合性能临界值的预测结果被纳入EM建模(图4)。对比4种指标的评价结果,所有单一模型的预测结果均显著优于随机分布模型,但不同单一模型的表现具有较大差异,这在AUC值的对比中最为明显[图4(1)]。此外,不同指标得出的模型准确性排序也有所不同,但有3种指标(AUC、KAPPA、OA)认为GBM和RF的预测表现优于其他单一模型[图4(1), (3), (4)],ANN和CTA则表现较差。所有指标均表明EM的预测准确性明显优于所有单一模型,且其预测性能也更加稳定,这在AUC的比较中更加明显[图4(1)]。
 图 4 8种单一模型和集成模型(Ensemble Model, EM)的准确性评价结果(1)AUC得分箱线图;(2)TSS得分箱线图;(3)KAPPA得分箱线图;(4)OA得分箱线图Figure 4. Model accuracy scores obtained by the eight single models and Ensemble Model (EM)(1) the box plot of the AUC scores; (2) the box plot of the TSS scores; (3) the box plot of the KAPPA scores; (4) the box plot of the OA scores
图 4 8种单一模型和集成模型(Ensemble Model, EM)的准确性评价结果(1)AUC得分箱线图;(2)TSS得分箱线图;(3)KAPPA得分箱线图;(4)OA得分箱线图Figure 4. Model accuracy scores obtained by the eight single models and Ensemble Model (EM)(1) the box plot of the AUC scores; (2) the box plot of the TSS scores; (3) the box plot of the KAPPA scores; (4) the box plot of the OA scores3.3 栖息地适宜性预测与准确性验证
各单一模型的栖息地预测结果存在差异(图5)。EM对龙头鱼的栖息地适宜性预测最为准确[图5(33)~(36)],GBM的预测表现和EM最为接近,而RF、GLM、ANN、CTA和SRE的适生区预测准确性存在明显的季节间差异[图5(5), (13), (15), (21), (25), (26), (31)]。以2015年为例,各单一模型间的栖息地适宜性高值区分布十分相似,EM能够识别模型间共同的适宜性高值区,且在几乎所有季节能够准确预测出龙头鱼的出现点和未出现点。相较于其余单一模型,EM对龙头鱼出现点所在区域能够识别出更高的适宜性,也能清晰地区分出未采样区域适宜性水平的差异,模拟出了龙头鱼栖息地适宜性分布的更多细节[图5(33)~(36)]。此外,大部分模型对龙头鱼未出现点的预测准确性低于其对出现点的预测准确性。例如在春季,大部分单一模型认为北支的内部水域适宜性同样较高[图5(1), (9), (13), (21), (25)],但该水域的调查站位并未监测到龙头鱼,即使是EM,其在该站点的预测结果也和观测结果并不一致[图5(33)]。根据EM预测结果,长江口龙头鱼的适生区范围自春季至秋季逐渐扩大,且主要分布在长江口北支以及北支口外的连续海域[图5(33~36)],冬季时研究区域内的适生区范围达到最低[图5(36)]。
 图 5 2015年8种单一模型和集成模型(Ensemble Model, EM)预测的龙头鱼适生区分布与实际调查结果的对比(1)~(4)GBM各季节预测结果;(5)~(8)RF各季节预测结果;(9)~(12)FDA各季节预测结果;(13)~(16)GLM各季节预测结果;(17)~(20)GAM各季节预测结果;(21)~(24)ANN各季节预测结果;(25)~(28)CTA各季节预测结果;(29)~(32)SRE各季节预测结果;(33)~(36)EM各季节预测结果Figure 5. Comparison of the distribution of suitable habitat for Harpadon nehereus predicted by the eight single models and Ensemble Model (EM) in 2015 with the actual observations(1) − (4) seasonal predictions of GBM; (5) − (8) seasonal predictions of RF; (9) − (12) seasonal predictions of FDA; (13) − (16) seasonal predictions of GLM; (17) − (20) seasonal predictions of GAM; (21) − (24) seasonal predictions of ANN; (25) − (28) seasonal predictions of CTA; (29) − (32) seasonal predictions of SRE; (33) − (36) seasonal predictions of EM
图 5 2015年8种单一模型和集成模型(Ensemble Model, EM)预测的龙头鱼适生区分布与实际调查结果的对比(1)~(4)GBM各季节预测结果;(5)~(8)RF各季节预测结果;(9)~(12)FDA各季节预测结果;(13)~(16)GLM各季节预测结果;(17)~(20)GAM各季节预测结果;(21)~(24)ANN各季节预测结果;(25)~(28)CTA各季节预测结果;(29)~(32)SRE各季节预测结果;(33)~(36)EM各季节预测结果Figure 5. Comparison of the distribution of suitable habitat for Harpadon nehereus predicted by the eight single models and Ensemble Model (EM) in 2015 with the actual observations(1) − (4) seasonal predictions of GBM; (5) − (8) seasonal predictions of RF; (9) − (12) seasonal predictions of FDA; (13) − (16) seasonal predictions of GLM; (17) − (20) seasonal predictions of GAM; (21) − (24) seasonal predictions of ANN; (25) − (28) seasonal predictions of CTA; (29) − (32) seasonal predictions of SRE; (33) − (36) seasonal predictions of EM3.4 解释变量重要性分析
3.4.1 生物和非生物性变量重要性
本研究在4种算法中选择最具代表性的5个模型进行变量重要性分析(图6)。EM模拟结果代表了所选模型的平均趋势,各环境变量的重要性从大到小依次为:Sal,Tem,COD,prey,Chl a,pH,Depth[图6(6)],其中Sal和Tem对龙头鱼栖息地适宜性的贡献率显著高于其他所有变量。6个模型中,有4个模型(GBM、RF、CTA、EM)将Sal识别为贡献率最高的环境变量[图6—(1), (2), (3), (6)],4个模型(GBM、RF、CTA、EM)将Tem识别为次要环境变量[图6(1), (2), (3), (6)]。几乎所有模型认为prey、Chl a、pH和Depth的重要性明显低于上述关键栖息地需求因子,SRE的次要环境变量识别结果与其他模型不同,但其对于关键需求因子的识别基本与其他所选模型一致[图6(5)]。
 图 6 5种单一模型和集成模型(Ensemble Model, EM)预测的各环境变量重要性雷达图(Tem:水温;Chl a:叶绿素a;Sal:盐度;COD:化学需氧量;Depth:水深;prey:饵料生物资源密度)(1)GBM的因子重要性雷达图;(2)RF的因子重要性雷达图;(3)CTA的因子重要性雷达图;(4)GAM的因子重要性雷达图;(5)SRE的因子重要性雷达图;(6)EM的因子重要性雷达图Figure 6. Radar charts of the importance of each environmental variable predicted by the five single models and Ensemble Model (EM) (Tem: temperature; Chl a: chlorophyll a; Sal: salinity; COD: chemical oxygen demand; prey: the stock density of prey species)(1) radar chart of the importance of variables in GBM; (2) radar chart of the importance of variables in RF; (3) radar chart of the importance of variables in CTA; (4) radar chart of the importance of variables in GAM; (5) radar chart of the importance of variables in SRE; (6) radar chart of the importance of variables in EM
图 6 5种单一模型和集成模型(Ensemble Model, EM)预测的各环境变量重要性雷达图(Tem:水温;Chl a:叶绿素a;Sal:盐度;COD:化学需氧量;Depth:水深;prey:饵料生物资源密度)(1)GBM的因子重要性雷达图;(2)RF的因子重要性雷达图;(3)CTA的因子重要性雷达图;(4)GAM的因子重要性雷达图;(5)SRE的因子重要性雷达图;(6)EM的因子重要性雷达图Figure 6. Radar charts of the importance of each environmental variable predicted by the five single models and Ensemble Model (EM) (Tem: temperature; Chl a: chlorophyll a; Sal: salinity; COD: chemical oxygen demand; prey: the stock density of prey species)(1) radar chart of the importance of variables in GBM; (2) radar chart of the importance of variables in RF; (3) radar chart of the importance of variables in CTA; (4) radar chart of the importance of variables in GAM; (5) radar chart of the importance of variables in SRE; (6) radar chart of the importance of variables in EM3.4.2 关键环境变量的偏依赖关系比较
根据变量重要性分析结果,选择Sal、Tem和COD绘制变量偏依赖曲线图(图7)。由于SRE仅产生二元预测结果,参考价值较低,因此不做展示。其余所选单一模型能够预测出相似的变化趋势,但得分相对较差的单一模型如CTA的偏依赖曲线表现得非常“钝化”,而EM明显绘制出了优势模型的集中趋势。EM指出在Sal、Tem、COD范围分别为2.754~30.300[图7(1)]、28.278~30.934℃[图7(2)]、4.605~8.080 mg/L[图7(3)]时,属于龙头鱼的高适宜性栖息地。
 图 7 4种单一模型和集成模型(Ensemble Model, EM)绘制的环境变量偏依赖图(Sal:盐度;Tem:水温;COD:化学需氧量)(1)Sal的偏依赖图;(2)Tem的偏依赖图;(3)COD的偏依赖图Figure 7. Partial dependence plots of environmental variables obtained by the four single models and Ensemble Model (EM) (Sal: salinity; Tem: temperature; COD: chemical oxygen demand)(1) partial dependence plot of Sal; (2) partial dependence plot of Tem; (3) partial dependence plot of COD
图 7 4种单一模型和集成模型(Ensemble Model, EM)绘制的环境变量偏依赖图(Sal:盐度;Tem:水温;COD:化学需氧量)(1)Sal的偏依赖图;(2)Tem的偏依赖图;(3)COD的偏依赖图Figure 7. Partial dependence plots of environmental variables obtained by the four single models and Ensemble Model (EM) (Sal: salinity; Tem: temperature; COD: chemical oxygen demand)(1) partial dependence plot of Sal; (2) partial dependence plot of Tem; (3) partial dependence plot of COD4. 讨论
4.1 单一模型的适用性和局限性
机器学习模型的有效性已被多个研究证明,例如Elith等[31]比较了226个物种的16种建模方法,发现类似于机器学习的方法优于其他成熟方法;Phillips等[37]开发了GLM、FDA、RF等6种方法对翻车鲀(Mola mola)和拉氏翻车鲀(Mola ramsayi)的栖息地分布进行建模分析,认为机器学习方法可以产生比剖面和统计方法更优秀的预测性能。理论上,基于机器学习算法的模型结构是根据数据确定的,能够避免模型规范所需的假设[38]。另外,上述模型的建模方法本身就已经包含了集成技术,模型能够更灵活地分析物种栖息地适宜性和环境变量之间的空间异质性非线性关系,从而使得物种自身属性和特征对模型的影响较小,以有效减少模型过度拟合[39]。具体到长江口,该水域内栖息地类型复杂镶嵌,且龙头鱼物种发生(出现)数据具有稀疏性特征,机器学习模型在避免过拟合和简单调整参数方面优于传统模型,因此能取得良好的预测准确性[40]。
由于建模技术的统计基础不同,模型即使使用相同的因变量和自变量,也可能产生完全不同的结论[41]。传统统计建模方法虽易于使用且具有良好的解释性,但现实生态位中需要模型考虑的环境变量数据与物种发生(出现)数据间的关系具有强烈的非线性和非稳态特征[42],从而使得传统建模方法的模型特征无法准确契合生态现实。
当然,也有研究发现具有较高统计学准确性的机器学习模型因为不能提供清晰的统计学原理,而可能增加过拟合等风险[43]。由此可见,没有一种模型的预测性能能够始终保持最优,进行广泛的模型性能比较或者使用集成方法可能成为了选择合适SDMs方法的主要方式。
4.2 集成方法对长江口龙头鱼SDMs表现的改进
先前的研究发现集成方法无需通过模型对比以选择“最佳”单一模型,而且可产生更准确的预测结果。例如,杨晓龙等[44]使用10种单一模型开发出1种加权EM,以生成山东省天鹅湖大叶藻(Zostera marina)的高分辨率适宜性生境地图,发现加权EM的栖息地预测准确性优于所有单一模型;Stohlgren等[45]测试了5种单一模型和1种EM在预测美国非本地植物物种的入侵分布方面的性能,发现EM比单一模型的预测性能更为稳健且准确。长江口龙头鱼的建模评估也支持EM具有最佳表现的观点,这可能是由于EM整合了多种SDMs的输出结果,能够识别每种单一模型带有的物种发生和环境变量关系的真实“信号”,并分离由数据和模型结构中的误差和不确定性造成的“噪声”[46]。
此外,EM能够识别出不同模型共同的高适宜区域,并清晰地区分出未采样区域适宜性水平的差异,为栖息地保护工作提供了更可靠的决策依据、更完善的调查计划以及更明确的保护顺序。以2015年秋季为例,EM和GBM、RF、FDA等模型共同将长江口北支至北支口外的连续海域识别为适宜性高值区,说明优先在上述区域开展栖息地保护工作的风险较小,这一结论符合观测数据的验证结果[图5(3), (7), (11), (15), (35)]。在春季,EM将北支入海口外西北−东南向狭长水域也识别为龙头鱼的高适宜性栖息地,区分出更多未采样区域适宜性水平的差异[图5(33)],说明今后可能需要进一步完善调查设计,增加对长江口龙头鱼栖息地使用模式的了解。EM在保护规划中的有效性可以通过平均函数的低通滤波能力来解释[47],基于加权平均法的EM具有“清洗”效应,预测结果可以避免出现由于单一模型预测值过高或过低而造成的孤立适生和非适生区,从而为栖息地保护规划工作提供更可靠的科学依据。
4.3 基于集成方法的长江口龙头鱼关键栖息地需求识别
识别物种的关键栖息地需求是除栖息地适宜性预测以外SDMs的另一项关键输出,由于符合筛选标准的单一模型具有较为接近的识别结果,为长江口龙头鱼构建的EM所揭示的变量重要性和偏依赖关系均体现了多模型的集中趋势,近似于上述单一模型的混合(图6(6)、 图7)。因此,在关键栖息地需求的识别方面,EM避免了随机选择单一模型可能会面临的风险,为理解物种的栖息地偏好提供了更安全和稳健的决策基础。例如,在对水温的偏依赖关系识别中,性能评价中表现良好的GAM只能识别出适宜性峰值所在范围,无法提供更详细的响应关系信息,可能将导致栖息地保护决策偏离龙头鱼的生态位特征[图7(2)]。
根据EM的预测结果,盐度和水温对龙头鱼在长江口的分布发挥着关键影响。龙头鱼成鱼倾向于在繁殖期到来之前洄游至高盐度水域以预备产卵,而龙头鱼仔稚鱼和幼鱼对盐度的耐受性虽然较成鱼更低,但其仍然能够大量栖息于盐度小于27的环境[48]。水温变化将直接影响龙头鱼的摄食、生长、繁殖和新陈代谢,从而进一步作用于龙头鱼的洄游活动[49]。
5. 不足与展望
本研究的结果证实了EM能够有效提高预测结果和观测结果的一致性,减少了利用SDMs预测海洋物种栖息地中基于模型类型的不确定性。然而,准确可靠地预测河口性鱼类物种的分布仍面临着诸多挑战。渔业资源监测数据的采样偏差、物种所在位置的不准确记录和数据集较小也可能对SDMs的准确性产生很大影响[50]。因此,在后续研究中也应持续关注SDMs预测结果不确定性的其他来源,继续减少模型结果的误差,为长江口龙头鱼和其他重要水生生物资源的管理、保护和可持续利用提供更有效的科学工具。
-
图 2 构建长江口龙头鱼栖息地模型的技术路线图(GBM:梯度提升机;RF:随机森林;ANN:人工神经网络;CTA:分类树分析;FDA:柔性判别分析;GAM:广义加性模型;GLM:广义线性模型;SRE:表面分布区分室模型;EM:集成模型)
Fig. 2 Technical framework for constructing the habitat model of Harpadon nehereus in the Changjiang River Estuary (GBM: Gradient Boosting Machine; RF: Random Forest; ANN: Artificial Neural Network; CTA: Classification Tree Analysis; FDA: Flexible Discriminant Analysis; GAM: Generalized Additive Model; GLM: Generalized Linear Model; SRE: Surface Range Envelop; EM: Ensemble Model)
图 3 各解释变量的皮尔逊相关性系数矩阵分析图(COD:化学需氧量;Depth:水深;Tem:水温;Chl a:叶绿素a;Sal:盐度;DO:溶解氧;prey:饵料生物资源密度)
Fig. 3 Pearson correlation coefficient for explanatory variables (COD: chemical oxygen demand; Tem: temperature; Chl a: chlorophyll a; Sal: salinity; DO: dissolved oxygen; prey: the stock density of prey species)
图 4 8种单一模型和集成模型(Ensemble Model, EM)的准确性评价结果
(1)AUC得分箱线图;(2)TSS得分箱线图;(3)KAPPA得分箱线图;(4)OA得分箱线图
Fig. 4 Model accuracy scores obtained by the eight single models and Ensemble Model (EM)
(1) the box plot of the AUC scores; (2) the box plot of the TSS scores; (3) the box plot of the KAPPA scores; (4) the box plot of the OA scores
图 5 2015年8种单一模型和集成模型(Ensemble Model, EM)预测的龙头鱼适生区分布与实际调查结果的对比
(1)~(4)GBM各季节预测结果;(5)~(8)RF各季节预测结果;(9)~(12)FDA各季节预测结果;(13)~(16)GLM各季节预测结果;(17)~(20)GAM各季节预测结果;(21)~(24)ANN各季节预测结果;(25)~(28)CTA各季节预测结果;(29)~(32)SRE各季节预测结果;(33)~(36)EM各季节预测结果
Fig. 5 Comparison of the distribution of suitable habitat for Harpadon nehereus predicted by the eight single models and Ensemble Model (EM) in 2015 with the actual observations
(1) − (4) seasonal predictions of GBM; (5) − (8) seasonal predictions of RF; (9) − (12) seasonal predictions of FDA; (13) − (16) seasonal predictions of GLM; (17) − (20) seasonal predictions of GAM; (21) − (24) seasonal predictions of ANN; (25) − (28) seasonal predictions of CTA; (29) − (32) seasonal predictions of SRE; (33) − (36) seasonal predictions of EM
图 6 5种单一模型和集成模型(Ensemble Model, EM)预测的各环境变量重要性雷达图(Tem:水温;Chl a:叶绿素a;Sal:盐度;COD:化学需氧量;Depth:水深;prey:饵料生物资源密度)
(1)GBM的因子重要性雷达图;(2)RF的因子重要性雷达图;(3)CTA的因子重要性雷达图;(4)GAM的因子重要性雷达图;(5)SRE的因子重要性雷达图;(6)EM的因子重要性雷达图
Fig. 6 Radar charts of the importance of each environmental variable predicted by the five single models and Ensemble Model (EM) (Tem: temperature; Chl a: chlorophyll a; Sal: salinity; COD: chemical oxygen demand; prey: the stock density of prey species)
(1) radar chart of the importance of variables in GBM; (2) radar chart of the importance of variables in RF; (3) radar chart of the importance of variables in CTA; (4) radar chart of the importance of variables in GAM; (5) radar chart of the importance of variables in SRE; (6) radar chart of the importance of variables in EM
图 7 4种单一模型和集成模型(Ensemble Model, EM)绘制的环境变量偏依赖图(Sal:盐度;Tem:水温;COD:化学需氧量)
(1)Sal的偏依赖图;(2)Tem的偏依赖图;(3)COD的偏依赖图
Fig. 7 Partial dependence plots of environmental variables obtained by the four single models and Ensemble Model (EM) (Sal: salinity; Tem: temperature; COD: chemical oxygen demand)
(1) partial dependence plot of Sal; (2) partial dependence plot of Tem; (3) partial dependence plot of COD
表 1 本研究中各单一模型的参数设置
Tab. 1 Parameter settings for each single model in this study
类别 模型 参数 值 描述 机器学习算法 梯度提升机
(Gradient Boosting Machine, GBM)n.trees 2500 生成树的数量 interaction.depth 7 交互项的最大深度 shrinkage 0.001 在构建每棵树时,收缩节点的权重 随机森林
(Random forest, RF)ntree 500 生成树的数量 mtry default 每次分割时随机抽样作为候选变量的数量 nodesize 5 每个终端节点的最少样本数 人工神经网络
(Artificial Neural Network, ANN)size NULL 神经网络的规模 decay NULL 学习率衰减因子 maxit 200 训练神经网络时的最大迭代次数 分类算法 分类树分析
(Classification Tree Analysis, CTA)minbucket 5 节点中最少的样本数 minsplit 5 最小分裂点的值 cp 0.001 控制修剪范围 柔性判别分析(Flexible Discriminant Analysis, FDA) Method Mars FDA的建模方法 add_args NULL 附加参数列表 回归算法 广义加性模型(Generalized Additive Model, GAM) algo GAM_mgcv 使用的算法和优化方法 k −1 平滑器的阶次 interaction.level 0 交互项的阶次 广义线性模型
(Generalized Linear Model, GLM)type Quadratic 模型类型 interaction.level 0 交互项的阶次 mustart 0.5 默认起始值 包络算法 表面分布区分室模型(Surface Range Envelop, SRE) quant 0.025 模型的分辨率或离散程度 -
[1] 潘邵媛, 王学昉, 田思泉, 等. 长江口中华鲟保护区海洋环境监测浮标站点的优化设计[J]. 海洋学报, 2021, 43(4): 55−64.Pan Shaoyuan, Wang Xuefang, Tian Siquan, et al. The design of the stations of marine environmental monitoring buoys in the Chinese sturgeon nature reserve in the Changjiang River Estuary[J]. Haiyang Xuebao, 2021, 43(4): 55−64. [2] 杨钧渊, 陈锦辉, 钟俊生, 等. 长江口崇明东滩水域仔稚鱼种类组成及多样性[J]. 上海海洋大学学报, 2023, 32(4): 829−840. doi: 10.12024/jsou.20230204101Yang Junyuan, Chen Jinhui, Zhong Junsheng, et al. Species composition and diversity of fish larvae and juveniles in the water area of Chongming Dongtan, Yangtze River Estuary[J]. Journal of Shanghai Ocean University, 2023, 32(4): 829−840. doi: 10.12024/jsou.20230204101 [3] 潘绪伟, 程家骅. 长江口外海域龙头鱼营养生态学特征[J]. 中国水产科学, 2011, 18(5): 1132−1140.Pan Xuwei, Cheng Jiahua. Feeding ecology of Harpadon nehereus in areas adjacent to Changjiang River estuary[J]. Journal of Fishery Sciences of China, 2011, 18(5): 1132−1140. [4] Li Yu, Gao Chunxia, Chen Jinhui, et al. Spatial–temporal distribution characteristics of Harpadon nehereus in the Yangtze River Estuary and its relationship with environmental factors[J]. Frontiers in Marine Science, 2024, 11: 1340522. doi: 10.3389/fmars.2024.1340522 [5] 唐未, 王学昉, 吴峰, 等. 基于最大熵模型模拟西印度洋剑鱼栖息地的时空分布[J]. 海洋学报, 2022, 44(10): 100−108. doi: 10.12284/j.issn.0253-4193.2022.10.hyxb202210009Tang Wei, Wang Xuefang, Wu Feng, et al. Simulation of spatio-temporal distribution of swordfish habitat in the western Indian Ocean based on maximum entropy model[J]. Haiyang Xuebao, 2022, 44(10): 100−108. doi: 10.12284/j.issn.0253-4193.2022.10.hyxb202210009 [6] Le Pape O, Baulier L, Cloarec A, et al. Habitat suitability for juvenile common sole (Solea solea, L. ) in the Bay of Biscay (France): A quantitative description using indicators based on epibenthic fauna[J]. Journal of Sea Research, 2007, 57(2/3): 126−136. [7] França S, Cabral H N. Predicting fish species distribution in estuaries: Influence of species’ ecology in model accuracy[J]. Estuarine, Coastal and Shelf Science, 2016, 180: 11−20. doi: 10.1016/j.ecss.2016.06.010 [8] Austin M. Species distribution models and ecological theory: A critical assessment and some possible new approaches[J]. Ecological Modelling, 2007, 200(1/2): 1−19. [9] Jiang Rijin, Sun Haoqi, Li Xiafang, et al. Habitat suitability evaluation of Harpadon nehereus in nearshore of Zhejiang province, China[J]. Frontiers in Marine Science, 2022, 9: 961735. doi: 10.3389/fmars.2022.961735 [10] Mohammadi A, Almasieh K, Nayeri D, et al. Comparison of habitat suitability and connectivity modelling for three carnivores of conservation concern in an Iranian montane landscape[J]. Landscape Ecology, 2022, 37(2): 411−430. doi: 10.1007/s10980-021-01386-5 [11] Poulos H M, Chernoff B, Fuller P L, et al. Ensemble forecasting of potential habitat for three invasive fishes[J]. Aquatic Invasions, 2012, 7(1): 59−72. doi: 10.3391/ai.2012.7.1.007 [12] 王寇, 李博, 李爱国, 等. 夏季长江口及其邻近海域湍流特征分析[J]. 海洋学报, 2021, 43(11): 22−31.Wang Kou, Li Bo, Li Aiguo, et al. Characteristics of turbulence in the Changjiang River Estuary and its adjacent waters in summer[J]. Haiyang Xuebao, 2021, 43(11): 22−31. [13] 史赟荣, 晁敏, 沈新强. 长江口张网鱼类群落结构特征及月相变化[J]. 海洋学报, 2014, 36(2): 81−92. doi: 10.3969/j.issn.0253-4193.2014.02.009Shi Yunrong, Chao Min, Shen Xinqiang. Characteristics and monthly variations of set net fish community structure in the Changjiang River estuary[J]. Haiyang Xuebao, 2014, 36(2): 81−92. doi: 10.3969/j.issn.0253-4193.2014.02.009 [14] 吴晓丹, 宋金明, 李学刚. 长江口邻近海域水团特征与影响范围的季节变化[J]. 海洋科学, 2014, 38(12): 110−119. doi: 10.11759/hykx20140305001Wu Xiaodan, Song Jinming, Li Xuegang. Ssonal variation of water mass characteristic and influence area in the Yangtze Estuary and its adjacent waters[J]. Marine Sciences, 2014, 38(12): 110−119. doi: 10.11759/hykx20140305001 [15] 周晓英. 长江口海域表层水温变化的气候特征[D]. 青岛: 中国海洋大学, 2005.Zhou Xiaoying. Climate characteristics of sea surface temperature (SST) variation in the Changjiang Estuary[D]. Qingdao: Ocean University of China, 2005. [16] 何柄震. 长江口海域营养物质时空演变趋势及其对入海通量的响应研究[D]. 沈阳: 沈阳大学, 2024.He Bingzhen. The spatiotemporal evolution trend of nutrients in the Yangtze River Estuary and its response to the fluxes into the sea[D]. Shenyang: Shenyang University, 2024. [17] 朱宇新, 周斌, 赵俊杰. 盐水入侵对长江口盐度分布及影响研究[J]. 环境保护前沿, 2014, 4(1): 25−33. doi: 10.12677/AEP.2014.41B005Zhu Yuxin, Zhou Bin, Zhao Junjie. Research on the salinity distribution and the influence of saltwater intrusion in Yangtze River[J]. Advances in Environmental Protection, 2014, 4(1): 25−33. doi: 10.12677/AEP.2014.41B005 [18] 马金, 黄金玲, 陈锦辉, 等. 基于GAM的长江口鱼类资源时空分布及影响因素[J]. 水产学报, 2020, 44(6): 958−968.Ma Jin, Huang Jinling, Chen Jinhui, et al. Analysis of spatiotemporal fish density distribution and its influential factors based on generalized additive model (GAM) in the Yangtze River estuary[J]. Journal of Fisheries of China, 2020, 44(6): 958−968. [19] Wang Yichuan, Wu Xinghua, Zheng Leifu, et al. Modeling seasonal changes in the habitat suitability of Coilia nasus in the Yangtze River Estuary using tree-based methods[J]. Regional Studies in Marine Science, 2023, 67: 103212. doi: 10.1016/j.rsma.2023.103212 [20] França S, Costa M J, Cabral H N. Inter- and intra-estuarine fish assemblage variability patterns along the Portuguese coast[J]. Estuarine, Coastal and Shelf Science, 2011, 91(2): 262−271. doi: 10.1016/j.ecss.2010.10.035 [21] 中华人民共和国国家质量监督检验检疫总局, 中国国家标准化管理委员会. GB/T 12763.6−2007, 海洋调查规范 第6部分: 海洋生物调查[S]. 北京: 中国标准出版社, 2008: 8.General Administration of Quality Supervision, Inspection and Quarantine of the People's Republic of China, Standardization Administration of the People’s Republic of China. GB/T 12763.6−2007, Specifications for oceanographic survey-Part 6: Marine biological survey[S]. Beijing: Standards Press of China, 2008: 8. [22] 国家环境保护局. GB 11607−89, 渔业水质标准[S]. 北京: 中国标准出版社, 1990: 8.Environmental Protection Agency. GB 11607−89, Water quality standard for fisheries[S]. Beijing: Standards Press of China, 1990: 8. [23] 环境保护部. HJ 828—2017, 水质 化学需氧量的测定 重铬酸盐法[S]. 北京: 中国环境出版社, 2017: 3.Ministry of Environmental Protection. HJ 828—2017, Water quality-Determination of the chemical oxygen demand-Dichromate method[S]. Beijing: China Environmental Publishing House, 2017: 3. [24] Bacheler N M, Paramore L M, Buckel J A, et al. Abiotic and biotic factors influence the habitat use of an estuarine fish[J]. Marine Ecology Progress Series, 2009, 377: 263−277. doi: 10.3354/meps07805 [25] Luan Jing, Xu Binduo, Ji Yupeng, et al. Improving the spatial transferability of species distribution models to inform biological conservation of two piscivore fish species[J]. Biodiversity and Conservation, 2024, 33(14): 4215−4235. doi: 10.1007/s10531-024-02947-1 [26] 徐超, 王思凯, 赵峰, 等. 长江口水生动物食物网营养结构及其变化[J]. 水生生物学报, 2019, 43(1): 155−164. doi: 10.7541/2019.019Xu Chao, Wang Sikai, Zhao Feng, et al. Trophic structure of food web and its variation on aquatic animals in the Yangtze Estuary[J]. Acta Hydrobiologica Sinica, 2019, 43(1): 155−164. doi: 10.7541/2019.019 [27] Yalçın E, Gurbet R. Environmental influences on the spatio-temporal distribution of European Hake (Merluccius merluccius) in Izmir Bay, Aegean Sea[J]. Turkish Journal of Fisheries and Aquatic Sciences, 2016, 16(1): 1−14. [28] Dormann C F, Elith J, Bacher S, et al. Collinearity: A review of methods to deal with it and a simulation study evaluating their performance[J]. Ecography, 2013, 36(1): 27−46. doi: 10.1111/j.1600-0587.2012.07348.x [29] Shrestha U B, Sharma K P, Devkota A, et al. Potential impact of climate change on the distribution of six invasive alien plants in Nepal[J]. Ecological Indicators, 2018, 95: 99−107. doi: 10.1016/j.ecolind.2018.07.009 [30] Elith J, Graham C H, Anderson R P, et al. Novel methods improve prediction of species’ distributions from occurrence data[J]. Ecography, 2006, 29(2): 129−151. doi: 10.1111/j.2006.0906-7590.04596.x [31] Fielding A H, Bell J F. A review of methods for the assessment of prediction errors in conservation presence/absence models[J]. Environmental Conservation, 1997, 24(1): 38−49. doi: 10.1017/S0376892997000088 [32] Liu Canran, White M, Newell G. Measuring and comparing the accuracy of species distribution models with presence-absence data[J]. Ecography, 2011, 34(2): 232−243. doi: 10.1111/j.1600-0587.2010.06354.x [33] Hao Tianxiao, Elith J, Guillera-Arroita G, et al. A review of evidence about use and performance of species distribution modelling ensembles like BIOMOD[J]. Diversity and Distributions, 2019, 25(5): 839−852. doi: 10.1111/ddi.12892 [34] Marmion M, Parviainen M, Luoto M, et al. Evaluation of consensus methods in predictive species distribution modelling[J]. Diversity and Distributions, 2009, 15(1): 59−69. doi: 10.1111/j.1472-4642.2008.00491.x [35] Thuiller W, Georges D, Engler R. Biomod2: Ensemble platform for species distribution modelling[Z]. 2014. (查阅网上资料, 未找到本条文献出版信息, 请确认) [36] Liu Zunlei, Jin Yan, Yang Linlin, et al. Improving prediction for potential spawning areas from a two-step perspective: A comparison of multi-model approaches for sparse egg distribution[J]. Journal of Sea Research, 2024, 197: 102460. doi: 10.1016/j.seares.2023.102460 [37] Phillips N D, Reid N, Thys T, et al. Applying species distribution modelling to a data poor, pelagic fish complex: The ocean sunfishes[J]. Journal of Biogeography, 2017, 44(10): 2176−2187. doi: 10.1111/jbi.13033 [38] França S, Cabral H N. Predicting fish species richness in estuaries: Which modelling technique to use?[J]. Environmental Modelling & Software, 2015, 66: 17−26. [39] Dreiseitl S, Ohno-Machado L. Logistic regression and artificial neural network classification models: a methodology review[J]. Journal of Biomedical Informatics, 2002, 35(5/6): 352−359. [40] Li Zengguang, Ye Zhenjiang, Wan Rong, et al. Model selection between traditional and popular methods for standardizing catch rates of target species: A case study of Japanese Spanish mackerel in the gillnet fishery[J]. Fisheries Research, 2015, 161: 312−319. doi: 10.1016/j.fishres.2014.08.021 [41] 刘尊雷, 杨林林, 袁兴伟, 等. 基于集成模型的小黄鱼越冬群体适宜生境及其环境影响因素[J]. 应用生态学报, 2020, 31(6): 2076−2086.Liu Zunlei, Yang Linlin, Yuan Xingwei, et al. Overwintering distribution and its environmental determinants of small yellow croaker based on ensemble habitat suitability modeling[J]. Chinese Journal of Applied Ecology, 2020, 31(6): 2076−2086. [42] Povak N A, Hessburg P F, Reynolds K M, et al. Machine learning and hurdle models for improving regional predictions of stream water acid neutralizing capacity[J]. Water Resources Research, 2013, 49(6): 3531−3546. doi: 10.1002/wrcr.20308 [43] Dormann C F, Purschke O, Márquez J R G, et al. Components of uncertainty in species distribution analysis: a case study of the Great Grey Shrike[J]. Ecology, 2008, 89(12): 3371−3386. doi: 10.1890/07-1772.1 [44] Yang Xiaolong, Zhang Xiumei, Zhang Peidong, et al. Ensemble habitat suitability modeling for predicting optimal sites for eelgrass (Zostera marina) in the tidal lagoon ecosystem: Implications for restoration and conservation[J]. Journal of Environmental Management, 2023, 330: 117108. doi: 10.1016/j.jenvman.2022.117108 [45] Stohlgren T J, Ma P, Kumar S, et al. Ensemble habitat mapping of invasive plant species[J]. Risk Analysis, 2010, 30(2): 224−235. doi: 10.1111/j.1539-6924.2009.01343.x [46] Araújo M B, New M. Ensemble forecasting of species distributions[J]. Trends in Ecology & Evolution, 2007, 22(1): 42−47. [47] Araújo M B, Whittaker R J, Ladle R J, et al. Reducing uncertainty in projections of extinction risk from climate change[J]. Global Ecology and Biogeography, 2005, 14(6): 529−538. doi: 10.1111/j.1466-822X.2005.00182.x [48] 孙浩奇. 浙江近海龙头鱼生物学特征及其时空分布与环境因子关系[D]. 舟山: 浙江海洋大学, 2022.Sun Haoqi. A study on the biological characteristics, temporal and spatial distribution, and the relationship with environmental factors of Harpadon nehereus in Zhejiang Province coastal waters[D]. Zhoushan: Zhejiang Ocean University, 2022. [49] Lewin W C, Mehner T, Ritterbusch D, et al. The influence of anthropogenic shoreline changes on the littoral abundance of fish species in German lowland lakes varying in depth as determined by boosted regression trees[J]. Hydrobiologia, 2014, 724(1): 293−306. doi: 10.1007/s10750-013-1746-8 [50] Collins S D, Abbott J C, Mcintyre N E. Quantifying the degree of bias from using county-scale data in species distribution modeling: can increasing sample size or using county-averaged environmental data reduce distributional overprediction?[J]. Ecology and Evolution, 2017, 7(15): 6012−6022. doi: 10.1002/ece3.3115 -





 下载:
下载:
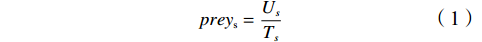
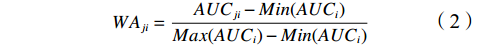
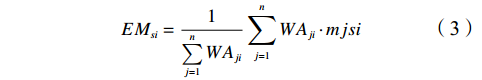

 下载:
下载:






