Effect of the content of colanic acid in marine bacterial biofilms on the settlement of Mytilus coruscus plantigrades
-
摘要: 可拉酸是生物被膜上重要的胞外多糖之一,但细菌可拉酸对海洋无脊椎动物附着过程的影响还鲜少研究。本研究从自然生物被膜中分离出8株海洋细菌,对其种属进行鉴定及聚类分析,并测定其生物被膜的可拉酸含量及对稚贝附着的诱导能力。筛选所得海洋细菌形成生物被膜并测定其成膜能力及胞外产物含量,发现β-多糖的生物量与厚壳贻贝稚贝附着率呈显著正相关趋势(p < 0.05)。8株海洋细菌生物被膜中可拉酸含量的定量结果显示,3株革兰氏阳性菌无法产生可拉酸,5株革兰氏阴性菌均可检测到不同含量的可拉酸,其中革兰氏阴性菌Shewanella marisflavi的可拉酸含量最高,为1 076.43 μg/mL。不同可拉酸含量的海洋细菌单一生物被膜与厚壳贻贝稚贝附着率之间关系的研究结果显示,海洋细菌生物被膜对厚壳贻贝稚贝附着率的诱导效果与其可拉酸含量呈显著正相关(p < 0.05)。以上结果表明,细菌生物被膜中的可拉酸能够参与诱导厚壳贻贝稚贝的附着。本研究为探究海洋细菌生物被膜的化学物质与海洋贝类附着之间的相互作用及其对贝类附着机制提出了新的见解。Abstract: Colanic acid is one of the vital exopolysaccharides in biofilms, yet the effect of marine bacterial colanic acid on the settlement of invertebrates is still rarely covered. In this study, eight strains of marine bacteria isolated from natural biofilms were identified and the phylogenetic analysis was carried out, the colanic acid content and inducing ability of biofilms were also determined. Before that, the biofilm formation capacity and the extracellular products of the screened bacteria were detected. It was found that β-polysaccharide had a significant positive correlation with the settlement rate of Mytilus coruscus plantigrades (p < 0.05). The quantitative results of colanic acid content showed that among the five Gram-negative bacteria which could produce colanic acid, Shewanella marisflavi had the highest colanic acid yield of 1076.43 μg/mL, and three Gram-positive bacteria could not generate it. The results of plantigrade settlement induced by different biofilms showed that the inducing activity of marine bacterial biofilms on the settlement rate of M. coruscus was positively correlated with the content of colanic acid (p < 0.05). These findings prove that the Gram-negative bacteria with colanic acid can positively regulate the settlement of M. coruscus plantigrades, while the Gram-positive bacteria without colanic acid have no inducing activity. This study provides further insights to figure out the interaction between the chemical cues of biofilms and marine molluscs.
-
Key words:
- biofilm /
- Mytilus coruscus /
- plantigrade /
- colanic acid /
- settlement
-
1. 引言
厚壳贻贝(Mytilus coruscus)广泛分布于东亚温带海域,是我国重要的商业化双壳贝类[1]。近年来,由于对该品种野生资源的掠夺性开发和规模化繁育技术不够成熟,导致该物种资源产量呈断崖式下跌,已难以满足市场需求[2]。为解决这一问题,节约生产成本并提高产量,对厚壳贻贝苗种繁育技术的探索显得尤为重要。与其他海洋无脊椎动物相似,厚壳贻贝的生长发育过程中会经历两个不同的生活阶段,其幼虫期经过附着变态过程后发育成稚贝,生活方式也因此由浮游生活状态转变为底栖附着,最终发育为成贝[3]。在厚壳贻贝稚贝生活阶段,外界环境条件的变化会使稚贝主动切断已经形成的足丝,寻找其他更为优质的附着基,并再次形成新的足丝以完成附着[4],这一生理过程被称为厚壳贻贝稚贝生活史中的二次附着。海区养殖方式中的贻贝脱苗问题是影响厚壳贻贝保苗率的重要因素,因此附着期稚贝的培育是厚壳贻贝人工育苗成功的关键[5],与贻贝养殖生产效益密切相关。
研究表明,细菌生物被膜广泛存在于自然海水中,生物被膜上化学信号及多种与凝集素结合的胞外蛋白等均可调控厚壳贻贝的附着[6-8]。多糖是细菌细胞表面物质的重要组成成分,对细胞的生存起着至关重要的作用[9]。在贻贝由幼虫转变为稚贝的过程中消耗了大量的能量[10],因而外界多糖的存在会对贻贝对附着基的选择过程产生一定的诱导作用[4, 11]。已有研究结果显示,胞外多糖的变化趋势与稚贝附着趋势呈现较高的一致性[11],推测多糖的含量变化是诱导稚贝附着的重要因素[12]。同时,细菌自身分泌的一些胞外多糖如可拉酸(Colanic Acid, CA)、纤维素等[13-15]均可以影响厚壳贻贝幼虫附着,2020年有研究首次提出厚壳贻贝幼虫的附着变态可能与可拉酸含量呈正相关[13],此后虽有研究表明可拉酸可以诱导幼虫附着,但对于可拉酸与稚贝附着之间的关系亟待研究。
1969年首次在大肠杆菌K235中发现可拉酸[16]。作为主要的细菌胞外多糖之一,可拉酸是一种高亲水性、高黏度的荚膜多糖,为白色纤维状物质[17],可溶于水或稀盐溶液[18],在多种细菌中存在,如:大肠杆菌(Escherichia coli)、迟缓爱德华氏菌(Edwardsiella tarda)、沙门氏菌(Salmonella)等[19-23]。由于其丰富的生物活性,可拉酸具有较高的商业价值。可拉酸可调控海洋生物的生长发育[13, 20]。有研究表明,纤维素的添加可以促进海假交替单胞菌(Pseudoalteromonas marina)产生可拉酸[24],从而诱导厚壳贻贝幼虫附着变态。迟缓爱德华氏菌可拉酸的缺失可以降低其对斑马鱼(Dario rerio)胚胎的毒力作用[20]。作为生物被膜成分,可拉酸有助于细菌抵抗噬菌体感染,并增强菌株对环境的应激反应[25]。有研究表明,可拉酸在细菌内的过度表达可能有助于沙门氏菌提高抗生素的抗性[26],有助于生物被膜的形成[27]。虽然有研究发现可拉酸可能分布在特定种属的细菌中[18],但海洋细菌中可拉酸的含量及其生物学功能还有待进一步研究。
本研究以自然生物被膜为来源,通过微生物培养技术获得多种海洋细菌的单一菌株,建立系统发育树分析其亲缘关系;分析不同海洋细菌的成膜能力及其胞外产物含量,分析探究胞外产物与稚贝附着之间的相关性;探究不同海洋细菌的可拉酸产生能力及其生物被膜对厚壳贻贝稚贝的诱导活性。本实验旨在发掘海洋细菌中可拉酸含量与厚壳贻贝稚贝附着的深层关系,为探究可拉酸与厚壳贻贝稚贝附着之间的相互作用机制提供数据支撑及理论依据。
2. 材料与方法
2.1 实验材料
本研究使用的野生厚壳贻贝稚贝(壳长:(1.67 ± 0.17)mm,壳高:(1.24 ± 0.15)mm)均来源于浙江省嵊泗县枸杞岛(30°46'N,122°44'E)。培养条件:海水盐度恒定30,每2 d定期定时换水,每日投喂青岛大扁藻(Platymonas helgolandica tsingtaoensis),在实验室18℃环境中避光充气暂养至少1周。
用于纯种海洋细菌分离纯化的自然微生物被膜源自浙江省舟山市嵊泗县海域,方法为Zobell 2216E稀释涂布法及平板划线法[28]。
2.2 实验方法
2.2.1 海洋细菌的分离纯化
自然生物被膜上海洋细菌的分离纯化方法参考Bao等[29]的方法。将自然生物被膜全部刮取后收集于1.5 mL微量离心管中,温度条件为4℃时,以800 r/min的转速离心,弃上清,液氮冷冻后于−80℃冷冻保存。使用时提前冰上融化,加入200 μL灭菌过滤海水(Autoclaved Filtered Seawater,AFSW)作为原液使用,等浓度梯度稀释至10−6。由原液至10−6的等浓度梯度稀释样液涂布培养48 h。分离单菌落纯化培养,重复多次纯化步骤以得到纯种菌株。于Zobell 2216E液体培养基(5 g蛋白胨、1 g酵母膏和0.01 g柠檬酸铁,调整pH范围在7.6~7.8之间)中扩大培养,扩培后的菌液作为菌种原液与60%甘油−NaCl溶液1∶1混合,−80℃冰箱中冷冻保存。
2.2.2 海洋细菌16S rDNA基因测序鉴定
使用天根生化科技有限公司的细菌基因组DNA提取试剂盒(DP302)提取各海洋细菌DNA。使用16S rDNA序列扩增特异性引物扩增后,由生工生物工程(上海)股份有限公司进行扩增产物测序分析。
2.2.3 生物被膜制备
取冻存的海洋细菌菌种活化后转接于100 mL Zobell 2216E液体培养基中,25℃恒温培养箱中震荡培养16~18 h。培养后海洋细菌菌株以3 500 r/min转速离心15 min,尽可能充分保留沉淀。菌体用AFSW重悬后离心,重复此步骤至上清液澄清。向彻底去除培养基的菌体加入新的50 mL AFSW,计算细菌密度。将菌液分别按相应初始菌浓度要求加至无菌玻璃培养皿中,培养皿中提前放入灭菌载玻片(38 mm × 26 mm)。AFSW定容至皿中终体积为20 mL。18℃条件下避光培养48 h,不同菌株不同浓度分别设置9个平行。
2.2.4 生物被膜胞外产物激光扫描共聚焦显微镜CLSM拍摄及图像处理
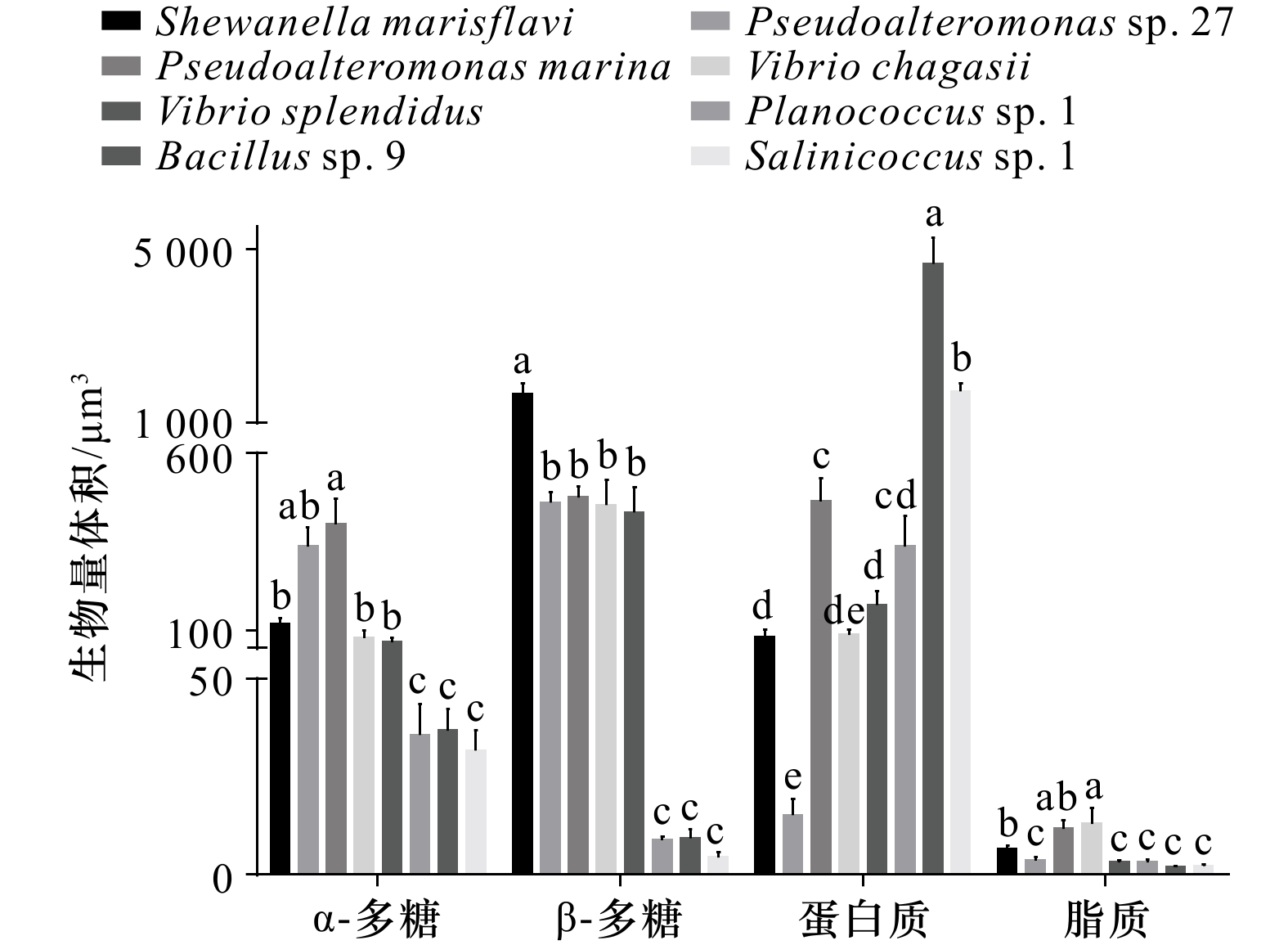
染色方法参考文献[30],染料成分及染色对象见表1。将生物被膜用浓度为5%的福尔马林溶液固定24 h后,在避光条件下用5 μg/mL的碘化丙啶(Propidium Iodide, PI)进行染色15~20 min,在激光扫描共聚焦显微镜(Confocal Laser Scanning Microscope,CLSM)(徕卡,德国) 630×放大倍率下进行观察拍摄。每组生物被膜分别选取3个载玻片,每个载玻片上拍摄3个视野,即每组载玻片共扫描拍摄9个视野进行成像,观察并对胞外产物生物量数据比较分析。每幅CLSM图像采用LAS X版本采集(像素为1 024 × 1 024,z-step为0.20 μm)。采用Image J软件对各组分进行定量分析。利用CLSM图像的阈值计算各组分的生物体积(单位:μm3)
表 1 激光扫描共聚焦显微镜荧光染料信息Table 1. Confocal laser scanning microscope fluorescence dye information染料 结合物质 波长/nm 碘化丙啶(PI) 死细菌 561 四甲基罗丹明共轭物刀豆蛋白( ConA-TMR) α-多糖 552/578 卡尔科弗卢尔荧光增白剂(CFW) β-多糖 254/432 1,1'−双十八烷基−3,3,3',3'− 四甲基吲哚二碳
花菁高氯酸盐(DiD’oil)脂质 648/670 异硫氰酸荧光素(FITC) 蛋白质 495/519 2.2.5 可拉酸染色定性
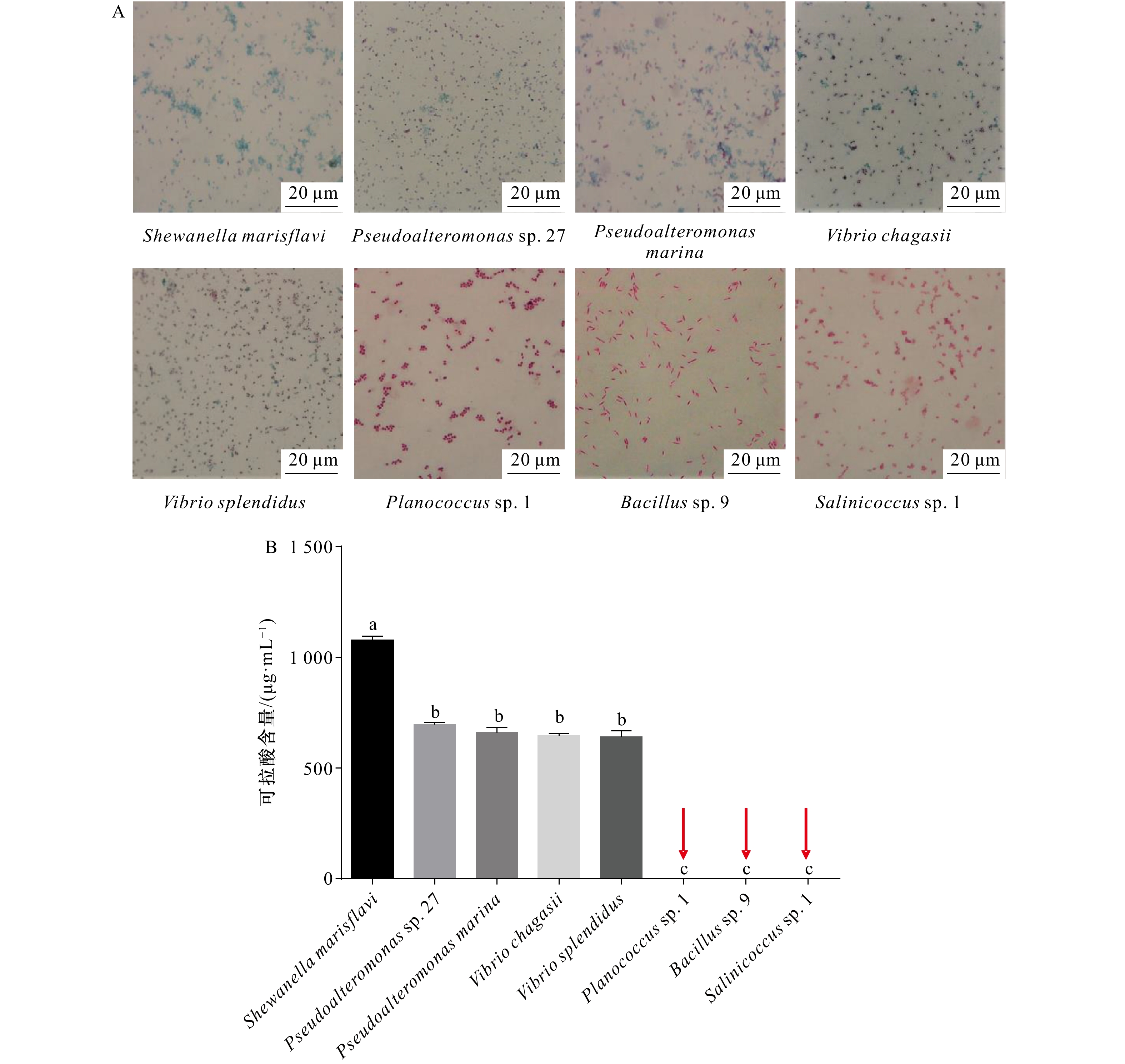
根据Ren等[31]的方法,各菌株成膜初始浓度设定为1×108 cell/mL。转移生物被膜至新的载玻片上,以品红溶液、媒染剂、1%亚甲基蓝溶液的染色顺序分别持续染色3 min、3 min、30 s。每次染色后的残余染料用蒸馏水轻轻浸润洗净。细菌在光学显微镜(目镜×10,物镜×100)下为红色,可拉酸呈蓝色。各实验组分别设置3个重复。
2.2.6 可拉酸含量测定根据
对Obadia 等[32]方法略加修改,以12片生物被膜为一组,每组收集至一个1.5 mL微量离心管中(n = 3)。加入1 mL双蒸水(double distilled H2O,ddH2O)将收集的生物被膜重悬,100℃加热10 min,13 000 rcf离心15~20 min,取含有可拉酸的上清液。1 mL上清液中加入4.5 mL H2SO4/H2O(6∶1)溶液,100℃条件下加热20 min,室温下冷却。取2 mL溶液在396 nm(A396-co)和427 nm(A427-co)下测吸光值。其余溶液分别加入100 μL 3%(m/v)半胱氨酸盐酸盐溶液,25℃避光静置1 h,396 nm(A396-cy)和427 nm(A427-cy)处测吸光值。以L-岩藻糖(Sigma)为浓度标准曲线。可拉酸含量用公式:(A396cy−A396co)−(A427cy−A427co)来计算。
2.2.7 厚壳贻贝稚贝附着
将培养48 h后的生物被膜的载玻片转移至含有20 mL AFSW的无菌玻璃培养皿中,生物被膜转移后每个玻璃皿中各加入10只稚贝放于生物被膜周围。18℃恒温条件下避光暂养,于12 h、24 h、48 h及72 h时分别记录稚贝在细菌生物被膜上的附着情况。每株细菌的不同处理组分别有9个重复,对照组为空白玻璃载玻片。
2.2.8 细菌密度计数
参考文献[4],生物被膜使用5%的福尔马林固定液浸泡固定至少24 h,以0.1%吖啶橙(Acridine Orange)染色液避光条件下染色5 min,自然风干,荧光显微镜×1 000(物镜倍数×100,目镜倍数×10)视野下随机选取10个均匀视野,不同处理组分别设置3个重复。
2.2.9 数据处理
使用JMP 10.0.0、GraphPad Prism 6.01及IBM SPSS Statistics 25.0.0软件对各组实验数据分别进行相关性检验和差异性分析,r为相关性系数,p < 0.05时差异显著。
3. 结果与分析
3.1 海洋细菌的测序与定名
本实验从自然生物被膜中共筛选出8株海洋细菌,细菌特异性序列测序结果通过BLAST程序在NCBI数据库中比对,以确定细菌种属。结果如表2所示。
表 2 不同海洋细菌的16S rDNA序列分析Table 2. 16S rDNA sequence analysis of different marine bacteria序列号 登录号 对照菌名 比对序列号 相似度/% 来源 ECSMB14101 CP041153 Shewanella marisflavi CP041153 99.58 自然生物被膜 ECSMB14103 CP023558 Pseudoalteromonas marina CP023558 99.79 自然生物被膜 ECSMB14105 MT820512 Vibrio splendidus MT820512 99.93 自然生物被膜 ECSMB14107 CP034970 Vibrio chagasii MN938232 99.24 自然生物被膜 ECSMB20101 OP209745 Planococcus sp. 1 MH938046 99.93 自然生物被膜 ECSMB20104 OP209750 Pseudoalteromonas sp. 27 KX889969 99.93 自然生物被膜 ECSMB20107 OP209748 Bacillus sp. 9 MG309326 99.86 自然生物被膜 ECSMB21103 OP209747 Salinicoccus sp. 1 DQ001316 98.22 自然生物被膜 3.2 海洋细菌的表型及系统发育分析
观察筛选后的纯种单菌落形态及颜色(图1a),发现除两株球菌(Planococcus sp. 1,Salinicoccus sp. 1)形成菌落较小外,其余菌落形态特征上无明显差异。
 图 1 8株细菌的菌落形态及邻接法构建的系统发育树a. 8株细菌的表型特征; b. 邻接法构建16S rDNA基因序列系统发育树Figure 1. The colony morphological and phylogenetic tree constructed by the Neighbor-Joining method of the eight marine bacterial strainsa. The morphological characteristics of eight bacterial species; b. phylogenetic tree of 16S rDNA gene sequences constructed by the Neighbor-Joining method
图 1 8株细菌的菌落形态及邻接法构建的系统发育树a. 8株细菌的表型特征; b. 邻接法构建16S rDNA基因序列系统发育树Figure 1. The colony morphological and phylogenetic tree constructed by the Neighbor-Joining method of the eight marine bacterial strainsa. The morphological characteristics of eight bacterial species; b. phylogenetic tree of 16S rDNA gene sequences constructed by the Neighbor-Joining method通过邻接(Neighbor-Joining, NJ)法构建系统发育树(图1b),并计算菌株间的遗传距离(表3)。结果显示,Vibrio splendidus与Vibrio chagasii亲缘关系相近,遗传距离为0.036;Pseudoalteromonas sp. 27与 Pseudoalteromonas marina两株细菌间的遗传距离仅为0.021,可将其归于同一属;Bacillus sp.9与Planococcus sp.1遗传距离相对较近,为0.088,表明两者间的亲缘关系较近。而不同细菌属间亲缘关系相对较远。
表 3 8株海洋细菌遗传距离Table 3. Genetic distance of the eight marine bacterial strains测试菌株 Pseudoalteromonas
marinaPlanococcus
sp. 1Vibrio
splendidusVibrio
chagasiiSalinicoccus
sp. 1Bacillus
sp. 9Shewanella
marisflaviPseudoalteromonas
sp. 27Pseudoalteromonas marina 0 Planococcus sp. 1 0.262 0 Vibrio splendidus 0.126 0.259 0 Vibrio chagasii 0.139 0.267 0.036 0 Salinicoccus sp. 1 0.262 0.109 0.250 0.257 0 Bacillus sp. 9 0.256 0.088 0.263 0.261 0.113 0 Shewanella marisflavi 0.099 0.267 0.140 0.144 0.254 0.259 0 Pseudoalteromonas sp. 27 0.021 0.262 0.119 0.132 0.259 0.256 0.093 0 3.3 不同海洋细菌生物被膜诱导稚贝附着的能力
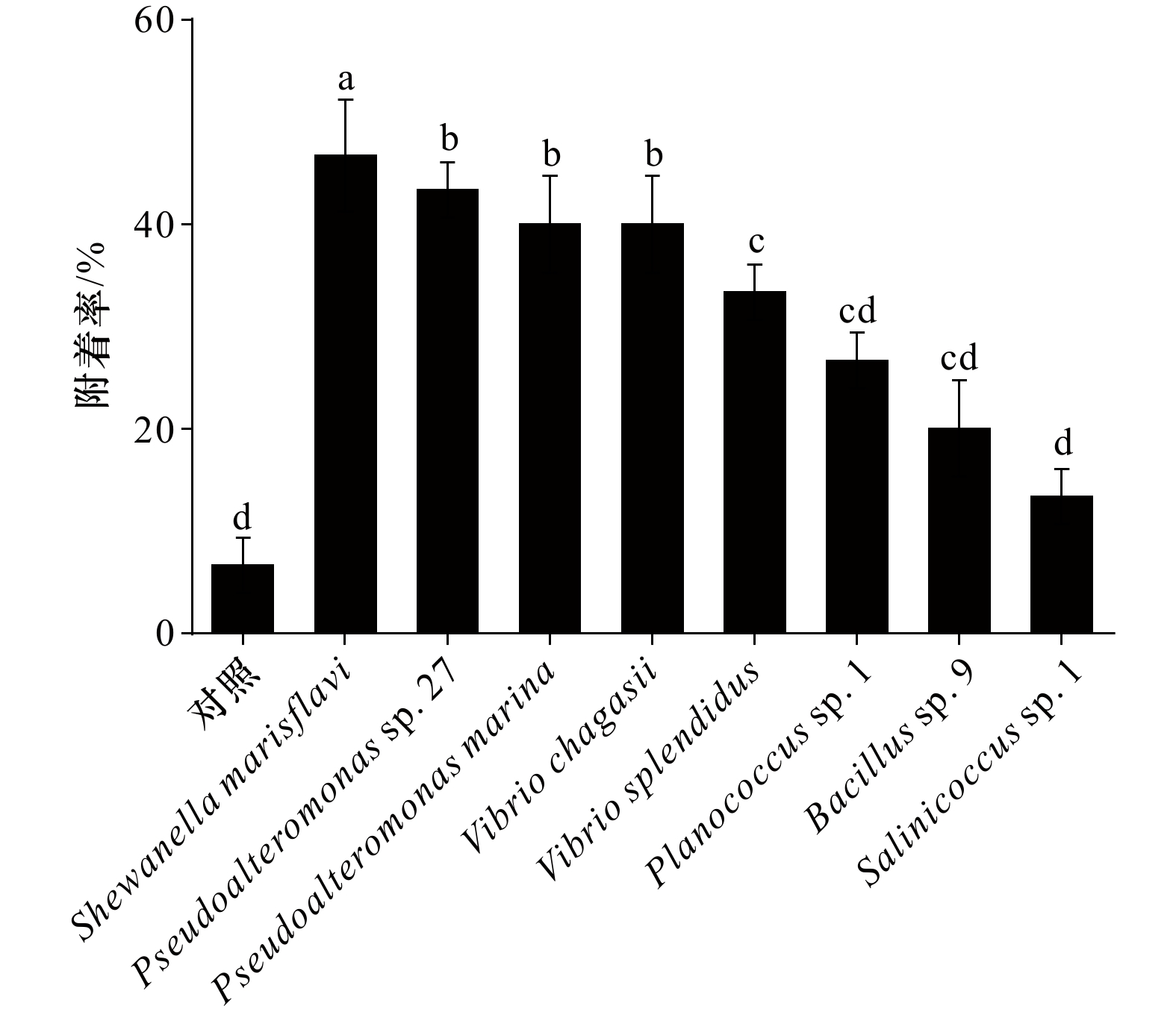
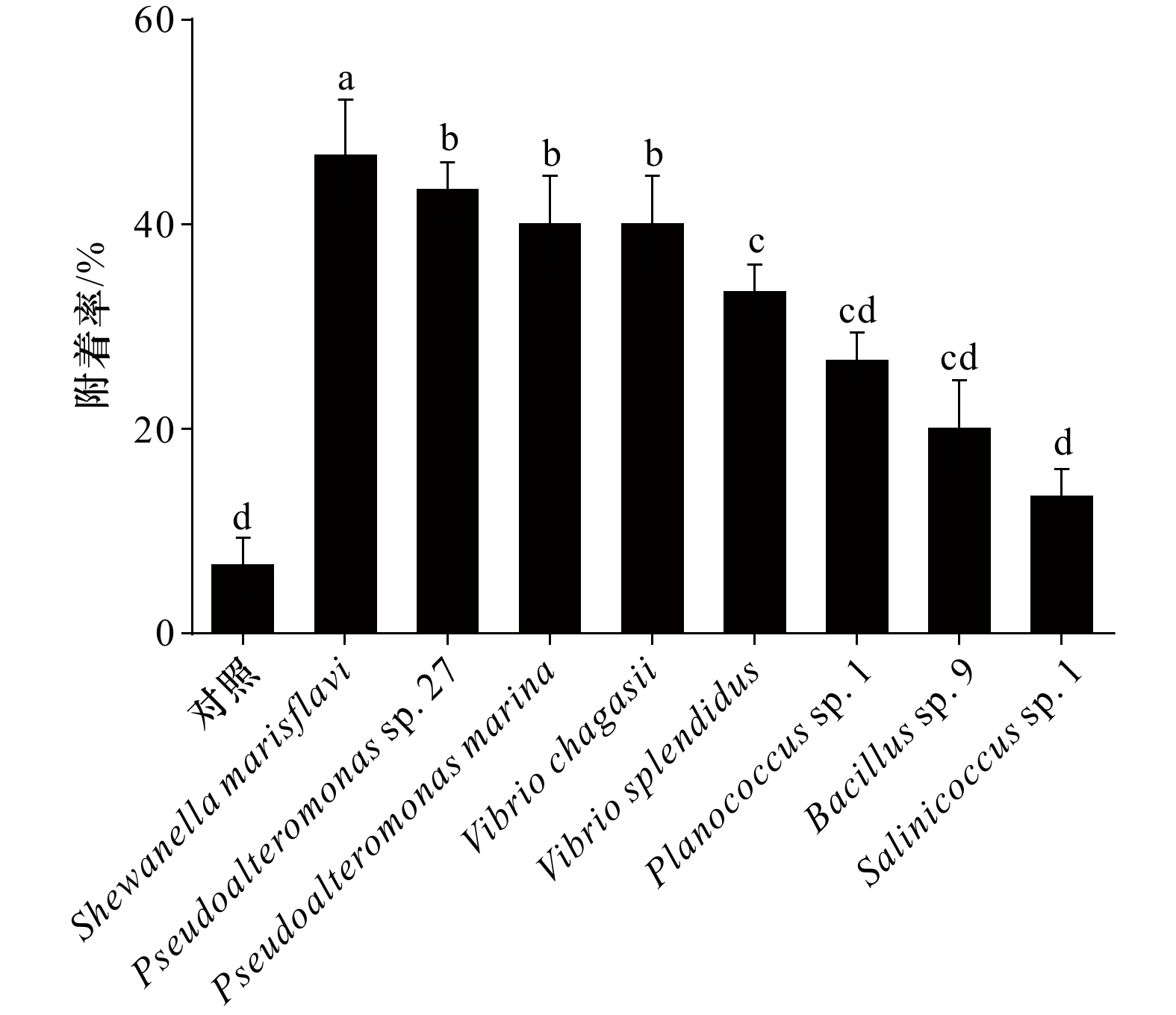
不同海洋细菌对厚壳贻贝稚贝附着的诱导效果如图2所示,空白对照组的稚贝附着率仅为(6.67± 3.79)%。相较于空白对照,Pseudoalteromonas sp. 27、Pseudoalteromonas marina、Vibrio splendidus、Vibrio chagasii、Shewanella marisflavi 均表现出明显的诱导活性(p < 0.05),其中Shewanella marisflavi 的诱导活性最高,为(46.67 ± 6.42)%;Salinicoccus sp. 1、Planococcus sp. 1、Bacillus sp. 9的诱导活性与空白对照间无显著性差异(p > 0.05),其中诱导活性最低的为Salinicoccus sp. 1((10.00 ± 0.10)%)。
3.4 不同海洋细菌生物被膜形成能力
生物被膜细菌终密度在一定程度上可以反应不同细菌生物被膜的成膜能力。如图3所示,8株海洋细菌显示出不同的成膜能力。不同海洋细菌的形态特征及其在膜上的分布状态如图3A所示,与其成膜后的细菌终密度所示一致(图3B)。当初始细菌密度为5 × 108 cell/cm2时,Shewanella marisflavi形成生物被膜后的终密度最高,与其他几株细菌的终密度呈显著差异(p < 0.05),为(3.3 × 108 ± 7.62 × 107)cell/cm2,但其生物被膜厚度却显著低于Pseudoalteromonas marina(图3C);其次为Pseudoalteromonas sp. 27,其成膜后细菌终密度为(4.87 × 107 ± 7.22 × 106)cell/cm2;Vibrio chagasii成膜后的细菌终密度为(6.26 × 106 ± 1.19 × 106)cell/cm2显著低于其他7株海洋细菌(p < 0.05),膜厚显著低于其他6株细菌,与Bacillus sp. 9相比,差异不显著,这表明Vibrio chagasii的成膜能力低于本文中的其他海洋细菌。
 图 3 不同海洋细菌生物被膜形成能力A. 不同海洋细菌形成生物被膜CLSM 拍摄图像;B. 不同海洋细菌形成生物被膜的终密度;C. 不同海洋细菌生物被膜膜厚;不同字母代表显著性差异Figure 3. Biofilm formation ability of different marine bacteriaA. Confocal laser scanning micrographs of biofilms; B. the density of eight monospecific bacterial biofilms; C. the thickness of biofilms; difference letters represent significant difference
图 3 不同海洋细菌生物被膜形成能力A. 不同海洋细菌形成生物被膜CLSM 拍摄图像;B. 不同海洋细菌形成生物被膜的终密度;C. 不同海洋细菌生物被膜膜厚;不同字母代表显著性差异Figure 3. Biofilm formation ability of different marine bacteriaA. Confocal laser scanning micrographs of biofilms; B. the density of eight monospecific bacterial biofilms; C. the thickness of biofilms; difference letters represent significant difference3.5 不同海洋细菌生物胞外产物分析
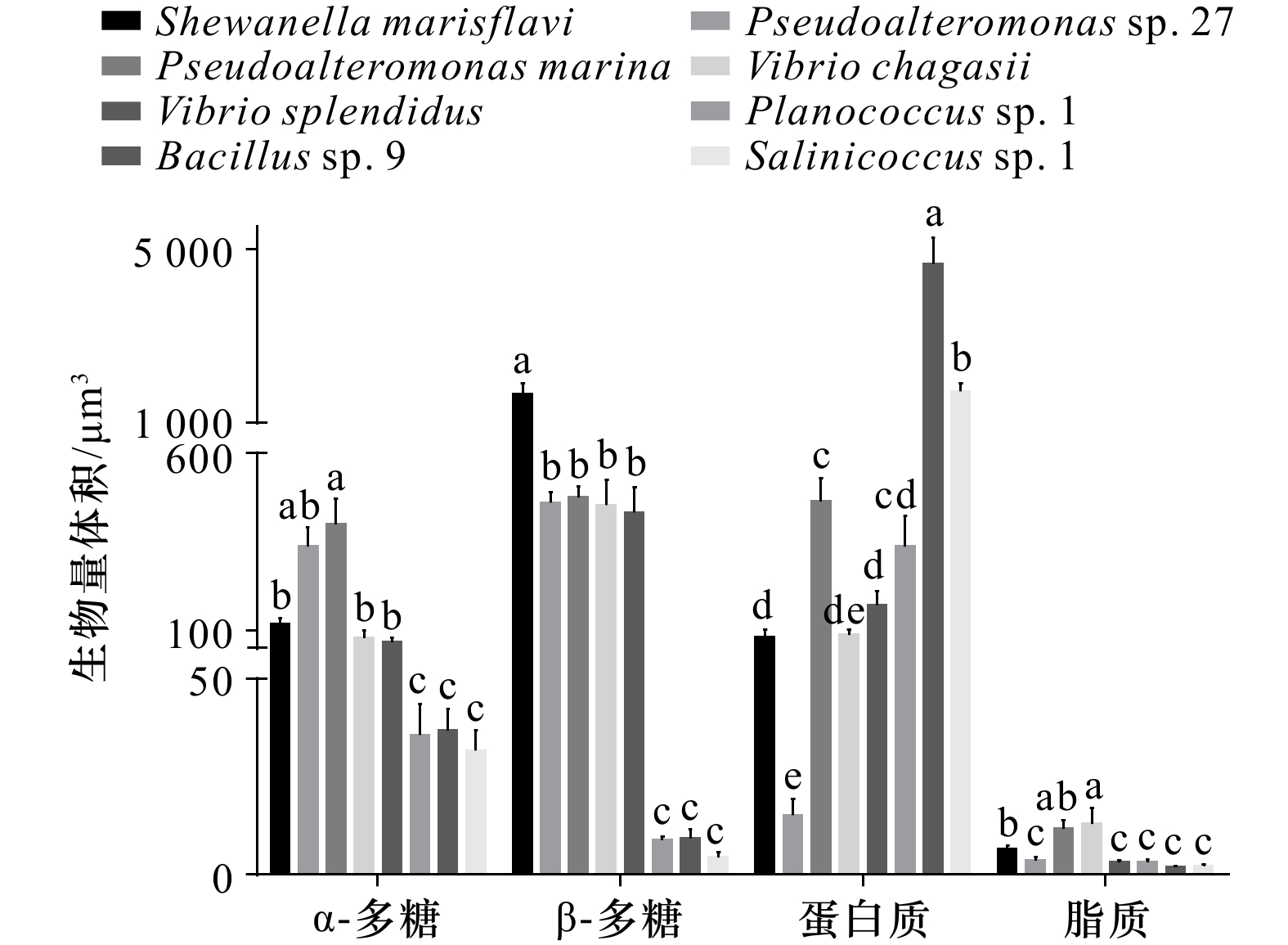
生物被膜组成成分十分复杂,为探究何种胞外产物在厚壳贻贝稚贝附着过程中发挥主要作用,本文通过CLSM对其胞外多糖、蛋白质及脂质进行定量分析(图A1),并利用CLSM图像的阈值计算各组分的生物量(图4),以此确定生物被膜各组分与稚贝附着之间的关系。如表4结果所示,β-多糖与稚贝附着之间呈现显著正相关(p < 0.05),稚贝附着率最高的Shwaneella marisflavi 其相对应的β-多糖的含量也最高,而α-多糖、蛋白质及脂质的含量与附着率之间未呈现相关性趋势。
表 4 胞外产物与厚壳贻贝稚贝附着率相关性分析Table 4. Correlation analyses between biofilm extracellular products and settlement rate of Mytilus coruscus plantigrades胞外产物 附着率 r p α-多糖 0.584 0.129 β-多糖 0.742 0.035* 蛋白质 −0.656 0.077 脂质 0.579 0.132 注:p为检验值;r为相关系数;*表示显著性差异(p < 0.05)。 3.6 8株海洋细菌的可拉酸含量分析
可拉酸作为重要的胞外多糖,其单糖组成结构中含有的α-多糖与β-多糖的比值为5∶1,为探究可拉酸是否发挥重要诱导作用,对8株海洋细菌的可拉酸含量进行了测定。如图5A所示,通过光学显微镜观察到不同海洋细菌的可拉酸生成能力不同。3株阳性细菌(Planococcus sp. 1,Bacillus sp. 9,Salinicoccus sp. 1)均不产生可拉酸,其余5株阴性细菌可产生可拉酸,但含量不同。图5B定量结果显示,3株阳性细菌中均未检测可拉酸,其余5株阴性菌种均可检测到不同含量的可拉酸。其中菌株Shewanella marisflavi 的可拉酸含量最高,达1076.43 μg/mL。
 图 5 8株海洋细菌的可拉酸染色(A)及可拉酸含量测定(B)A. 光镜下观察可拉酸在媒染生物膜上的分布,细菌被染成红色,可拉酸被染成蓝色;B. 生物被膜可拉酸浓度,红色箭头表示未检测到可拉酸,不同字母代表显著性差异Figure 5. The dyeing (A) and determination of the content (B) of colanic acid of eight marine pacteriaA. The distribution of colanic acid on mordant-stained biofilms under light microscope, the strains were stained red and the colanic acid was stained blue; B. the colanic acid concentration of biofilms, the red arrow indicates no colanic acid was detected, difference letters represent significant difference
图 5 8株海洋细菌的可拉酸染色(A)及可拉酸含量测定(B)A. 光镜下观察可拉酸在媒染生物膜上的分布,细菌被染成红色,可拉酸被染成蓝色;B. 生物被膜可拉酸浓度,红色箭头表示未检测到可拉酸,不同字母代表显著性差异Figure 5. The dyeing (A) and determination of the content (B) of colanic acid of eight marine pacteriaA. The distribution of colanic acid on mordant-stained biofilms under light microscope, the strains were stained red and the colanic acid was stained blue; B. the colanic acid concentration of biofilms, the red arrow indicates no colanic acid was detected, difference letters represent significant difference3.7 影响厚壳贻贝稚贝附着的多因素分析
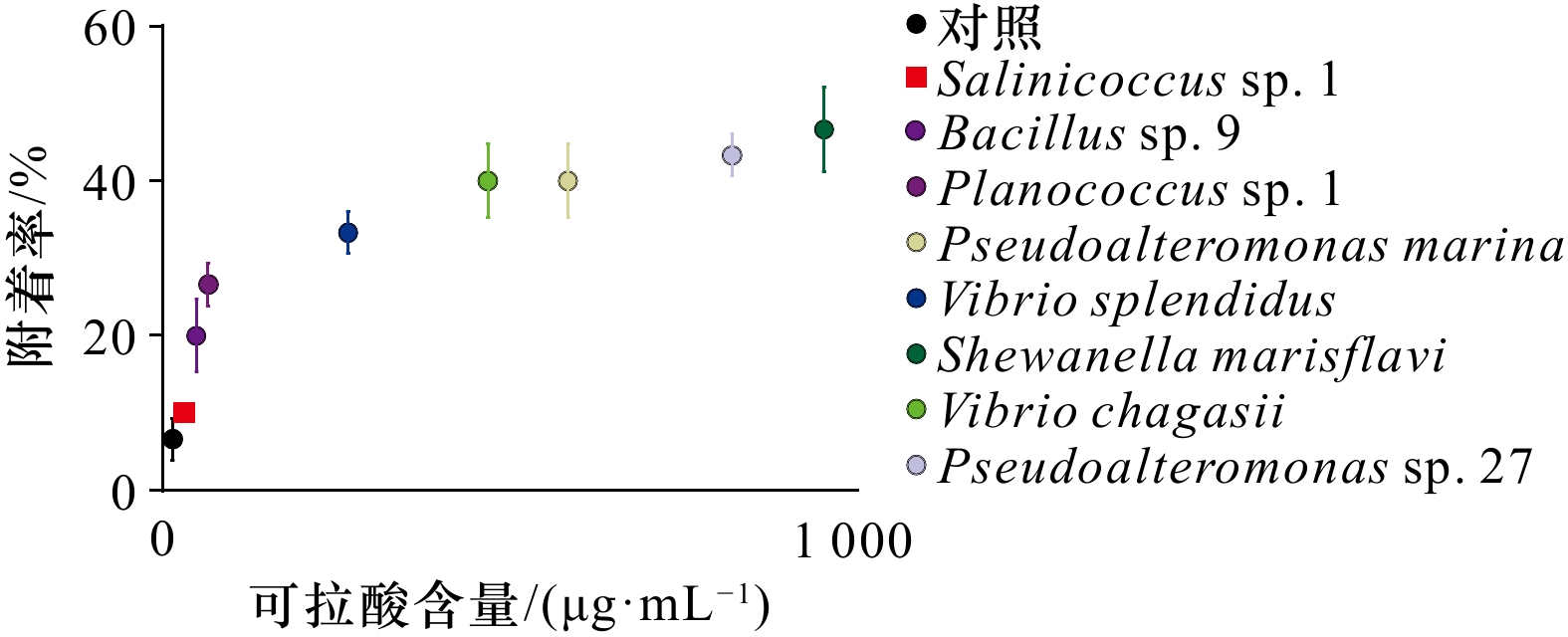
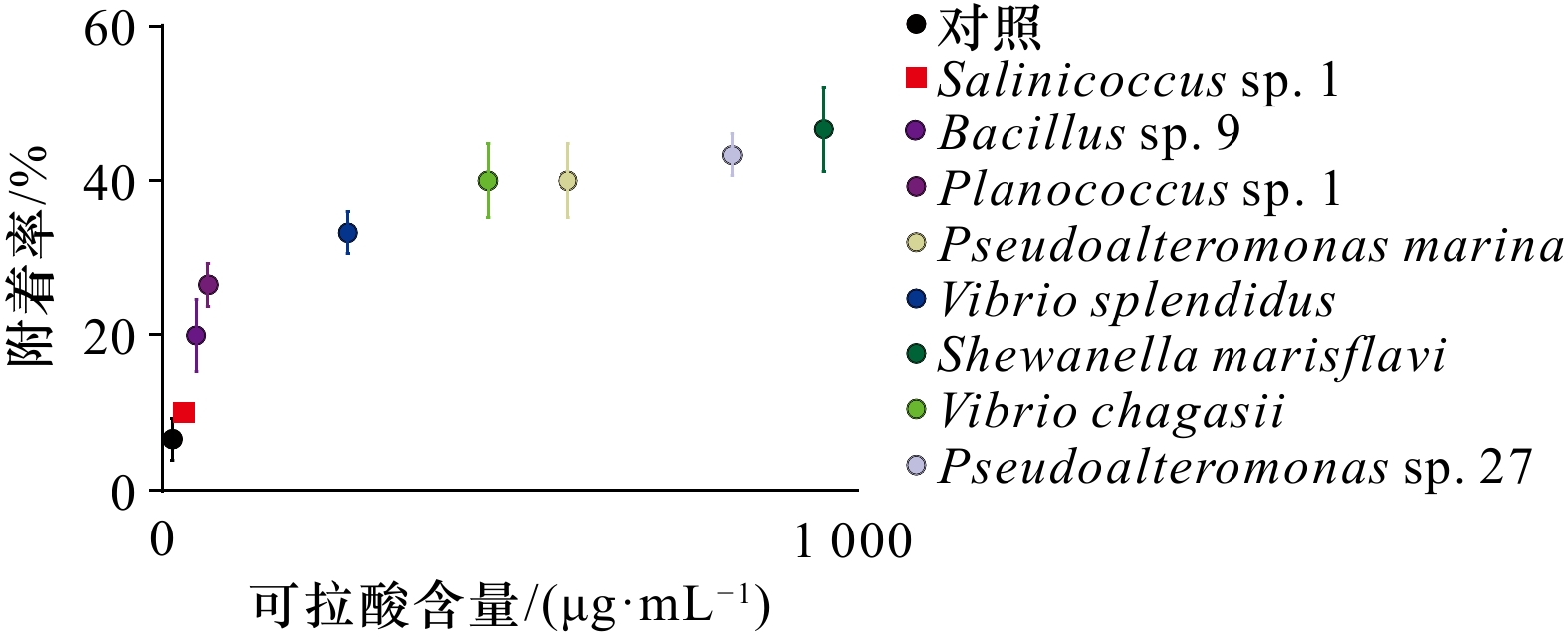
为确定可拉酸含量与厚壳贻贝稚贝附着的关系,本文选取定量检测可拉酸含量的相同初始细菌密度形成单一生物被膜进行稚贝附着实验,结果如图6所示。结果显示厚壳贻贝稚贝的附着率随可拉酸含量的升高而升高,其中,Shewanella marisflavi 的稚贝附着率最高,为46.67%。通过对8株不同种属细菌的细菌密度、可拉酸含量与厚壳贻贝稚贝附着率的相关性分析,发现可拉酸含量与稚贝附着率呈极显著相关(p < 0.001)。
4. 讨论
海洋细菌生物膜上的胞外多糖在诱导海洋贝类附着过程中起着至关重要的作用[9]。作为一种重要的胞外多糖,不同海洋细菌生物膜中的可拉酸含量及其对海洋贝类稚贝附着的诱导活性尚无研究。本研究从自然生物膜筛选出8株海洋细菌,通过分析可拉酸含量及其对稚贝附着诱导的作用,首次证明可拉酸对厚壳贻贝稚贝的附着过程具有正向诱导作用,为细菌与贝类的相互作用以及经济贝类的养殖研究提供了新的视角。
海产贝类在选择附着基的过程中受多种因子的调控[33],细菌种属的差异在一定程度上可以影响稚贝的附着过程[4]。以往的研究表明,由自然生物被膜上分离的海洋细菌所形成的单一生物被膜不同程度地影响海洋无脊椎动物幼虫[29, 34-35]及厚壳贻贝稚贝[28]的附着过程,其中Cobetia sp.菌呈现出较高的诱导活性,Pseudoalteromonas sp.中呈现出中低等诱导活性。此外,不同种属的细菌虽然都能诱导稚贝附着,但同一属的不同细菌在稚贝附着过程中也会起不同的诱导作用[36]。本研究从自然生物被膜上分离出的8株海洋细菌的诱导效果也不尽相同,其中5株革兰氏阴性菌对稚贝附着具有显著诱导效果,而3株革兰氏阳性菌无诱导活性。遗传距离及系统发育树所呈现的结果显示,Pseudoalteromonas sp. 27与 Pseudoalteromonas marina亲缘关系相近,Vibrio splendidus与Vibrio chagasii归为同一属。然而,研究结果发现,Pseudoalteromonas sp. 27 与 Pseudoalteromonas marina的稚贝附着率之间无差异,但Vibrio splendidus与Vibrio chagasii 的稚贝附着率存在显著差异。Vibrio chagasii与Pseudoalteromonas marina的遗传距离较大,但在稚贝附着的诱导作用中差异不显著。由此推测,细菌种属特异性与稚贝诱导效果之间无特定关联。
许多研究结果证明,细菌对贻贝附着的诱导活性与其生物被膜细菌终密度有关[29, 37-38]。此前的研究中发现,在筛选的细菌中5株细菌的生物被膜终密度与稚贝附着呈正相关,4株呈负相关,还有1株无相关性[36]。而本研究中,通过相关性分析发现,8株海洋细菌所形成生物被膜的细菌终密度与稚贝附着的诱导活性之间并无相关性(p < 0.05)。因此可以推测生物被膜细菌终密度可以在特定种属间对稚贝附着发挥一定的诱导效果,但并不是决定性因素。
由于生物被膜组成成分的复杂性[39],除细菌种属及细菌终密度的影响外,生物被膜上存在多种化学因子,如胞外多糖、胞外分泌蛋白及脂质等[40],这些化学信号均会在一定程度上影响厚壳贻贝稚贝附着。其中,胞外多糖作为生物膜胞外聚合物的主要成分[41],在调节稚贝附着中起着关键作用[11, 15]。同一菌株在其动态演替的过程中,在胞外蛋白及胞外脂质均无显著差异,而胞外多糖含量变化显著,并极大程度的影响了厚壳贻贝稚贝的附着[11]。可拉酸作为主要的胞外多糖之一,有研究表明,可拉酸含量的升高显著诱导了厚壳贻贝幼虫的附着变态行为[13]。基于上述研究,本研究对分离所得的海洋细菌生物被膜的胞外产物进行了含量测定,结果显示,β-多糖与细菌对厚壳贻贝稚贝附着的诱导能力呈正相关,与此前的研究结果一致。在可拉酸组成中,α-多糖与β-多糖比例为1 : 5[23, 42],因此本研究测定了分离所得的海洋细菌生物被膜中的可拉酸含量。结果表明,含可拉酸的细菌生物膜对稚贝附着的诱导活性显著高于不含可拉酸的细菌生物膜。同时,相关性分析结果显示,可拉酸含量的变化趋势与稚贝附着的诱导效应一致。这说明可拉酸对稚贝附着有正向调控作用。
研究表明,贻贝的附着过程需要消耗一定的能量[43],影响其能量代谢的因素还尚不明确。众所周知,线粒体是一种高度动态的细胞器[44],参与生物体的能量代谢。由于线粒体可以在多种能量代谢中起关键作用,它可以改变细胞的代谢状态,影响生物机体的多种生理过程[45]。2017年Han等[46]发现肠道菌群中可拉酸的含量可以调控秀丽隐杆线虫(Caenorhabditis elegans )的寿命,过量的可拉酸可减少线粒体断裂,并促进其裂变,以此来延缓宿主的衰老过程,首次提出可拉酸的过量表达是一种连接微生物代谢和寄主寿命的益生菌机制[46]。此外,研究团队还证明了可拉酸对线粒体的调控作用具有保守性[46]。本研究发现,稚贝附着率与可拉酸含量呈正相关,因此推测,稚贝的线粒体可能是可拉酸的靶向位点,通过促进线粒体的不断裂变,使碎片化的线粒体更易被招募到需要ATP的细胞中[47],以此诱导稚贝向富含可拉酸的生物被膜附近移动,并促进稚贝足丝分泌等附着行为的发生,但可拉酸在稚贝附着过程中的具体作用机制仍需后续实验加以验证。
综上,不同种属的海洋细菌产生可拉酸的能力并不相同。相关分析首次证明了可拉酸含量与厚壳贻贝稚贝附着率的正相关关系。本研究结果为此后对厚壳贻贝稚贝附着机制的深入探究及厚壳贻贝的养殖育种提供了新的探究思路。
-
图 1 8株细菌的菌落形态及邻接法构建的系统发育树
a. 8株细菌的表型特征; b. 邻接法构建16S rDNA基因序列系统发育树
Fig. 1 The colony morphological and phylogenetic tree constructed by the Neighbor-Joining method of the eight marine bacterial strains
a. The morphological characteristics of eight bacterial species; b. phylogenetic tree of 16S rDNA gene sequences constructed by the Neighbor-Joining method
图 3 不同海洋细菌生物被膜形成能力
A. 不同海洋细菌形成生物被膜CLSM 拍摄图像;B. 不同海洋细菌形成生物被膜的终密度;C. 不同海洋细菌生物被膜膜厚;不同字母代表显著性差异
Fig. 3 Biofilm formation ability of different marine bacteria
A. Confocal laser scanning micrographs of biofilms; B. the density of eight monospecific bacterial biofilms; C. the thickness of biofilms; difference letters represent significant difference
图 5 8株海洋细菌的可拉酸染色(A)及可拉酸含量测定(B)
A. 光镜下观察可拉酸在媒染生物膜上的分布,细菌被染成红色,可拉酸被染成蓝色;B. 生物被膜可拉酸浓度,红色箭头表示未检测到可拉酸,不同字母代表显著性差异
Fig. 5 The dyeing (A) and determination of the content (B) of colanic acid of eight marine pacteria
A. The distribution of colanic acid on mordant-stained biofilms under light microscope, the strains were stained red and the colanic acid was stained blue; B. the colanic acid concentration of biofilms, the red arrow indicates no colanic acid was detected, difference letters represent significant difference
表 1 激光扫描共聚焦显微镜荧光染料信息
Tab. 1 Confocal laser scanning microscope fluorescence dye information
染料 结合物质 波长/nm 碘化丙啶(PI) 死细菌 561 四甲基罗丹明共轭物刀豆蛋白( ConA-TMR) α-多糖 552/578 卡尔科弗卢尔荧光增白剂(CFW) β-多糖 254/432 1,1'−双十八烷基−3,3,3',3'− 四甲基吲哚二碳
花菁高氯酸盐(DiD’oil)脂质 648/670 异硫氰酸荧光素(FITC) 蛋白质 495/519 表 2 不同海洋细菌的16S rDNA序列分析
Tab. 2 16S rDNA sequence analysis of different marine bacteria
序列号 登录号 对照菌名 比对序列号 相似度/% 来源 ECSMB14101 CP041153 Shewanella marisflavi CP041153 99.58 自然生物被膜 ECSMB14103 CP023558 Pseudoalteromonas marina CP023558 99.79 自然生物被膜 ECSMB14105 MT820512 Vibrio splendidus MT820512 99.93 自然生物被膜 ECSMB14107 CP034970 Vibrio chagasii MN938232 99.24 自然生物被膜 ECSMB20101 OP209745 Planococcus sp. 1 MH938046 99.93 自然生物被膜 ECSMB20104 OP209750 Pseudoalteromonas sp. 27 KX889969 99.93 自然生物被膜 ECSMB20107 OP209748 Bacillus sp. 9 MG309326 99.86 自然生物被膜 ECSMB21103 OP209747 Salinicoccus sp. 1 DQ001316 98.22 自然生物被膜 表 3 8株海洋细菌遗传距离
Tab. 3 Genetic distance of the eight marine bacterial strains
测试菌株 Pseudoalteromonas
marinaPlanococcus
sp. 1Vibrio
splendidusVibrio
chagasiiSalinicoccus
sp. 1Bacillus
sp. 9Shewanella
marisflaviPseudoalteromonas
sp. 27Pseudoalteromonas marina 0 Planococcus sp. 1 0.262 0 Vibrio splendidus 0.126 0.259 0 Vibrio chagasii 0.139 0.267 0.036 0 Salinicoccus sp. 1 0.262 0.109 0.250 0.257 0 Bacillus sp. 9 0.256 0.088 0.263 0.261 0.113 0 Shewanella marisflavi 0.099 0.267 0.140 0.144 0.254 0.259 0 Pseudoalteromonas sp. 27 0.021 0.262 0.119 0.132 0.259 0.256 0.093 0 表 4 胞外产物与厚壳贻贝稚贝附着率相关性分析
Tab. 4 Correlation analyses between biofilm extracellular products and settlement rate of Mytilus coruscus plantigrades
胞外产物 附着率 r p α-多糖 0.584 0.129 β-多糖 0.742 0.035* 蛋白质 −0.656 0.077 脂质 0.579 0.132 注:p为检验值;r为相关系数;*表示显著性差异(p < 0.05)。 -
[1] 常亚青. 贝类增养殖学[M]. 北京: 中国农业出版社, 2007.Chang Yaqing. Mollusc Culture[M]. Beijing: China Agriculture Press, 2007. [2] 常抗美, 刘慧慧, 李家乐, 等. 紫贻贝和厚壳贻贝杂交及F1代杂交优势初探[J]. 水产学报, 2008, 32(4): 552−557.Chang Kangmei, Liu Huihui, Li Jiale, et al. A primary study on hybridization of Mytilus galloprovincialis, Mytilus coruscus, heterosis of F1 generation[J]. Journal of Fisheries of China, 2008, 32(4): 552−557. [3] Castro P, Huber M E. Marine Biology[M]. 10th ed. New York: McGraw-Hill Education, 2016. [4] 杨金龙, 郭行磐, 陈芋如, 等. 中湿度表面的海洋细菌对厚壳贻贝稚贝附着的影响[J]. 水产学报, 2015, 39(3): 421−428.Yang Jinlong, Guo Xingpan, Chen Yuru, et al. Effects of bacterial biofilms formed on middle wettability surfaces on settlement of plantigrades of the mussel Mytilus coruscus[J]. Journal of Fisheries of China, 2015, 39(3): 421−428. [5] 李慷均, 顾成柏. 厚壳贻贝的北方人工繁育技术[J]. 水产养殖, 2015, 36(8): 35−37.Li Kangjun, Gu Chengbai. Artificial breeding techniques of Mytilus coruscus in northern China. [J]. Journal of Aquaculture, 2015, 36(8): 35−37. [6] Yang Jinlong, Li Xiang, Liang Xiao, et al. Effects of natural biofilms on settlement of plantigrades of the mussel Mytilus coruscus[J]. Aquaculture, 2014, 424−425: 228−233. doi: 10.1016/j.aquaculture.2014.01.007 [7] Li Yifeng, Guo Xingpan, Yang Jinlong, et al. Effects of bacterial biofilms on settlement of plantigrades of the mussel Mytilus coruscus[J]. Aquaculture, 2014, 433: 434−441. doi: 10.1016/j.aquaculture.2014.06.031 [8] Li Yifeng, Chen Yuru, Yang Jinlong, et al. Effects of substratum type on bacterial community structure in biofilms in relation to settlement of plantigrades of the mussel Mytilus coruscus[J]. International Biodeterioration & Biodegradation, 2014, 96: 41−49. [9] Limoli D H, Jones C J, Wozniak D J. Bacterial extracellular polysaccharides in biofilm formation and function[J]. Microbiology Spectrum, 2015, 3(3): 223−247. [10] Sánchez-Lazo C, Martínez-Pita I. Biochemical and energy dynamics during larval development of the mussel Mytilus galloprovincialis (Lamarck, 1819)[J]. Aquaculture, 2012, 358−359: 71−78. doi: 10.1016/j.aquaculture.2012.06.021 [11] 梁箫, 刘红雨, 杨丽婷, 等. 弧菌生物被膜的动态演替对厚壳贻贝附着的影响[J]. 水产学报, 2020, 44(1): 118−129.Liang Xiao, Liu Hongyu, Yang Liting, et al. Effects of dynamic succession of Vibrio biofilms on settlement of the mussel Mytilus coruscus[J]. Journal of Fisheries of China, 2020, 44(1): 118−129. [12] Zeng Zhenshun, Guo Xingpan, Li Baiyuan, et al. Characterization of self-generated variants in Pseudoalteromonas lipolytica biofilm with increased antifouling activities[J]. Applied Microbiology and Biotechnology, 2015, 99(23): 10127−10139. doi: 10.1007/s00253-015-6865-x [13] Peng Lihua, Liang Xiao, Chang Ruiheng, et al. A bacterial polysaccharide biosynthesis-related gene inversely regulates larval settlement and metamorphosis of Mytilus coruscus[J]. Biofouling, 2020, 36(7): 753−765. doi: 10.1080/08927014.2020.1807520 [14] Liang Xiao, Zhang Junbo, Shao Anqi, et al. Bacterial cellulose synthesis gene regulates cellular c-di-GMP that control biofilm formation and mussel larval settlement[J]. International Biodeterioration & Biodegradation, 2021, 165: 105330. [15] Peng Lihua, Liang Xiao, Xu Jiakang, et al. Monospecific biofilms of Pseudoalteromonas promote larval settlement and metamorphosis of Mytilus coruscus[J]. Scientific Reports, 2020, 10(1): 2577. doi: 10.1038/s41598-020-59506-1 [16] Goebel W F. Colanic acid[J]. Proceedings of the National Academy of Sciences of the United States of America, 1963, 49(4): 464−471. [17] Stevenson G, Andrianopoulos K, Hobbs M, et al. Organization of the Escherichia coli K-12 gene cluster responsible for production of the extracellular polysaccharide colanic acid[J]. Journal of Bacteriology, 1996, 178(16): 4885−4893. doi: 10.1128/jb.178.16.4885-4893.1996 [18] Grant W D, Sutherland I W, Wilkinson J F. Exopolysaccharide colanic acid and its occurrence in the Enterobacteriaceae[J]. Journal of Bacteriology, 1969, 100(3): 1187−1193. doi: 10.1128/jb.100.3.1187-1193.1969 [19] Shugar S, Sanderson K E. Characterization of a mucoid Escherichia coli K12 strain, and chemical analysis of the exopolysaccharide[J]. Canadian Journal of Microbiology, 1972, 18(7): 969−973. doi: 10.1139/m72-150 [20] Yu Min, Xu Ying, Xu Tingting, et al. WcaJ, the initiating enzyme for colanic acid synthesis, is required for lipopolysaccharide production, biofilm formation and virulence in Edwardsiella tarda[J]. Aquaculture, 2015, 437: 287−291. doi: 10.1016/j.aquaculture.2014.12.011 [21] Steenackers H, Hermans K, Vanderleyden J, et al. Salmonella biofilms: an overview on occurrence, structure, regulation and eradication[J]. Food Research International, 2012, 45(2): 502−531. doi: 10.1016/j.foodres.2011.01.038 [22] Raiger Iustman L J, Tribelli P M, Ibarra J G, et al. Genome sequence analysis of Pseudomonas extremaustralis provides new insights into environmental adaptability and extreme conditions resistance[J]. Extremophiles, 2015, 19(1): 207−220. doi: 10.1007/s00792-014-0700-7 [23] Zdorovenko E L, Kadykova A A, Shashkov A S, et al. Pantoea agglomerans P1a lipopolysaccharide: structure of the O-specific polysaccharide and lipid A and biological activity[J]. Carbohydrate Research, 2019, 484: 107767. doi: 10.1016/j.carres.2019.107767 [24] 梁箫, 童欢, 彭莉华, 等. 纤维素对海洋细菌生物被膜形成及厚壳贻贝幼虫附着变态的调控[J]. 大连海洋大学学报, 2020, 35(1): 75−82. doi: 10.16535/j.cnki.dlhyxb.2019-025Liang Xiao, Tong Huan, Peng Lihua, et al. Regulation of formation of biofilms and larval settlement and metamorphosis of mussel Mytilus coruscus by cellulose[J]. Journal of Dalian Ocean University, 2020, 35(1): 75−82. doi: 10.16535/j.cnki.dlhyxb.2019-025 [25] Chen Jinru, Lee S M, Mao Ying. Protective effect of exopolysaccharide colanic acid of Escherichia coli O157: H7 to osmotic and oxidative stress[J]. International Journal of Food Microbiology, 2004, 93(3): 281−286. doi: 10.1016/j.ijfoodmicro.2003.12.004 [26] Wang T K F, Yam W C, Yuen K Y, et al. Misidentification of a mucoid strain of Salmonella enterica serotype choleraesuis as Hafnia alvei by the Vitek GNI+ card system[J]. Journal of Clinical Microbiology, 2006, 44(12): 4605−4608. doi: 10.1128/JCM.01488-06 [27] Wang Chenhui, Zhang Hailing, Wang Jianli, et al. Colanic acid biosynthesis in Escherichia coli is dependent on lipopolysaccharide structure and glucose availability[J]. Microbiological Research, 2020, 239: 126527. doi: 10.1016/j.micres.2020.126527 [28] Yang Jinlong, Shen Peijing, Liang Xiao, et al. Larval settlement and metamorphosis of the mussel Mytilus coruscus in response to monospecific bacterial biofilms[J]. Biofouling, 2013, 29(3): 247−259. doi: 10.1080/08927014.2013.764412 [29] Bao Weiyang, Yang Jinlong, Satuito C G, et al. Larval metamorphosis of the mussel Mytilus galloprovincialis in response to Alteromonas sp. 1: evidence for two chemical cues?[J]. Marine Biology, 2007, 152(3): 657−666. doi: 10.1007/s00227-007-0720-2 [30] González-Machado C, Capita R, Riesco-Peláez F, et al. Visualization and quantification of the cellular and extracellular components of Salmonella Agona biofilms at different stages of development[J]. PLoS One, 2018, 13(7): e0200011. doi: 10.1371/journal.pone.0200011 [31] Ren Ge, Wang Zhou, Li Ye, et al. Effects of lipopolysaccharide core sugar deficiency on colanic acid biosynthesis in Escherichia coli[J]. Journal of Bacteriology, 2016, 198(11): 1576−1584. doi: 10.1128/JB.00094-16 [32] Obadia B, Lacour S, Doublet P, et al. Influence of tyrosine-kinase Wzc activity on colanic acid production in Escherichia coli K12 cells[J]. Journal of Molecular Biology, 2007, 367(1): 42−53. doi: 10.1016/j.jmb.2006.12.048 [33] Yang Jinlong, Li Yifeng, Liang Xiao, et al. Silver nanoparticles impact biofilm communities and mussel settlement[J]. Scientific Reports, 2016, 6: 37406. doi: 10.1038/srep37406 [34] Weiner R M, Segall A M, Colwell R R. Characterization of a marine bacterium associated with Crassostrea virginica (the eastern oyster)[J]. Applied and Environmental Microbiology, 1985, 49(1): 83−90. doi: 10.1128/aem.49.1.83-90.1985 [35] Lau S C K, Mak K K W, Chen Feng, et al. Bioactivity of bacterial strains isolated from marine biofilms in Hong Kong waters for the induction of larval settlement in the marine polychaete Hydroides elegans[J]. Marine Ecology Progress Series, 2002, 226: 301−310. doi: 10.3354/meps226301 [36] 周轩, 郭行磐, 陈芋如, 等. 低湿度表面的海洋附着细菌对厚壳贻贝附着的影响[J]. 大连海洋大学学报, 2015, 30(1): 30−35. doi: 10.3969/J.ISSN.2095-1388.2015.01.006Zhou Xuan, Guo Xingpan, Chen Yuru, et al. Effects of bacterial biofilms formed on low surface wettability on settlement of plantigrades of the mussel Mytilus coruscus[J]. Journal of Dalian Ocean University, 2015, 30(1): 30−35. doi: 10.3969/J.ISSN.2095-1388.2015.01.006 [37] Tran C, Hadfield M G. Larvae of Pocillopora damicornis (Anthozoa) settle and metamorphose in response to surface-biofilm bacteria[J]. Marine Ecology Progress Series, 2011, 433: 85−96. doi: 10.3354/meps09192 [38] Watnick P, Kolter R. Biofilm, city of microbes[J]. Journal of Bacteriology, 2000, 182(10): 2675−2679. doi: 10.1128/JB.182.10.2675-2679.2000 [39] Characklis W G, Cooksey K E. Biofilms and microbial fouling[J]. Advances in Applied Microbiology, 1983, 29: 93−138. [40] Flemming H C, Wingender J. The biofilm matrix[J]. Nature Reviews Microbiology, 2010, 8(9): 623−633. doi: 10.1038/nrmicro2415 [41] Seviour T, Derlon N, Dueholm M S, et al. Extracellular polymeric substances of biofilms: suffering from an identity crisis[J]. Water Research, 2019, 151: 1−7. doi: 10.1016/j.watres.2018.11.020 [42] Whitfield C. Biosynthesis and assembly of capsular polysaccharides in Escherichia coli[J]. Annual Review of Biochemistry, 2006, 75: 39−68. doi: 10.1146/annurev.biochem.75.103004.142545 [43] Dawirs R R, Dietrich A. Temperature and laboratory feeding rates in Carcinusmaenas L. (Decapoda: Portunidae) larvae from hatching through metamorphosis[J]. Journal of Experimental Marine Biology and Ecology, 1986, 99(2): 133−147. doi: 10.1016/0022-0981(86)90233-9 [44] Chan D C. Mitochondria: dynamic organelles in disease, aging, and development[J]. Cell, 2006, 125(7): 1241−1252. doi: 10.1016/j.cell.2006.06.010 [45] Yao Yanhua, Tsuchiyama S, Yang Ciyu, et al. Proteasomes, Sir2, and Hxk2 form an interconnected aging network that impinges on the AMPK/Snf1-regulated transcriptional repressor Mig1[J]. PLoS Genetics, 2015, 11(1): e1004968. doi: 10.1371/journal.pgen.1004968 [46] Han Bing, Sivaramakrishnan P, Lin C C J, et al. Microbial genetic composition tunes host longevity[J]. Cell, 2017, 169(7): 1249−1262.e13. doi: 10.1016/j.cell.2017.05.036 [47] Li Zheng, Okamoto K I, Hayashi Y, et al. The importance of dendritic mitochondria in the morphogenesis and plasticity of spines and synapses[J]. Cell, 2004, 119(6): 873−887. doi: 10.1016/j.cell.2004.11.003 期刊类型引用(4)
1. 张立杰,赵业,王浩娜,夏雨,张燕英,孙浩,许修明. 自然微生物膜对刺参幼体附着变态的影响. 烟台大学学报(自然科学与工程版). 2025(02): 177-185 .  百度学术
百度学术2. 马蕃,陶钰,杨金龙,梁箫. 海洋暖化对细菌生物被膜及其诱导厚壳贻贝幼虫变态能力的影响. 大连海洋大学学报. 2025(02): 241-249 .  百度学术
百度学术3. 徐筱琰,杜美荣,蒋增杰,姜娓娓,夏苏东,秦晓芳,严瑾,吕建福. 舟形藻生物膜诱导下虾夷扇贝(Mizuhopecten yessoensis)幼虫的变态及其蛋白质组学响应. 海洋与湖沼. 2024(04): 960-966 .  百度学术
百度学术4. 李浩然,何泽平,王钰怡,杨金龙,梁箫. 8株深渊细菌生物被膜特性及其对厚壳贻贝附着的作用. 大连海洋大学学报. 2024(06): 986-995 .  百度学术
百度学术其他类型引用(2)
-




 下载:
下载:






 下载:
下载:



 百度学术
百度学术